Diversidad y estructura genética de accesiones de palma de aceite (Elaeis guineesis Jacq.) provenientes de Camerún
Diversity and genetic structure of oil palm accession (Elaeis guineesis Jacq.) from Cameroon
DOI:
https://doi.org/10.15446/rev.colomb.biote.v16n2.40132Palabras clave:
Elaeis guineensis Jacq, marcadores microsatélite, alelos, flujo genético (es)Elaeis guineensis Jacq, microsatellite markers, alleles, gene flow (en)
https://doi.org/10.15446/rev.colomb.biote.v16n2.40132
ARTÍCULO DE INVESTIGACIÓN
Diversidad y estructura genética de accesiones de palma de aceite (Elaeis guineesis Jacq.)provenientes de Camerún
Diversity and genetic structure of oil palm accession (Elaeis guineesis Jacq.) from Cameroon
Estiben Pacheco1, Diana Arias2, Zaida Ojeda3, Hernán Romero4
1 BSc.Universidad Pedagógica y Tecnológica de Colombia, estiby88@hotmail.com
2 BSc., MSc. Investigador Asociado. Programa de Biología y Mejoramiento de la Palma. Corporación Centro de Investigación en Palma de Aceite - Cenipalma. dianaris2002@gmail.com
3 Lic., MSc. Docente Asistente. Escuela de Ciencias Biológicas. Facultad de Ciencias. Laboratorio de Biotecnología Vegetal BIOPLASMA. Universidad Pedagógica y Tecnológica de Colombia, sede Tunja. zaida.ojeda@uptc.edu.co
4 BSc., MSc., PhD. Coordinador del Programa de Biología y Mejoramiento de la Palma. Corporación Centro de Investigación en Palma de
Aceite - Cenipalma. Profesor Asociado. Departamento de Biología. Universidad Nacional de Colombia, sede Bogotá.
hromero@cenipalma.org
Recibido: diciembre 20 de 2013 Aprobado: octubre 20 de 2014
Resumen
La palma de aceite Elaeis guineesis Jacq. posee gran importancia debido al aceite que se extrae de sus frutos, del cual se obtienen derivados refinados de gran valor comercial como el biodiesel, entre otros. Esta investigación buscó determinar la estructura y diversidad genética de 311 muestras de palma de aceite proveniente de la República de Camerún mediante 10 marcadores microsatélite. Los resultados mostraron valores promedio para el número medio de alelos por locus de Na=8.433 y un número efectivo de alelos por locus de Ne=4.756; las diferencias entre estos valores permiten inferir que los 106 alelos encontrados para estas poblaciones son considerados alelos raros. Adicionalmente, el valor de diversidad genética fue alto (valor medio de He= 0.781) respecto a reportes de varios autores. La varianza molecular obtenida evidenció que el mayor porcentaje (80 %) se encuentra dentro de los individuos. Los análisis mostraron que no se definió ningún tipo de estructura poblacional, lo que permitió inferir un alto flujo genético entre las zonas geográficas, esto corroborado por los altos valores de diversidad genética obtenidos. Los 311 genotipos evaluados fueron definidos como una población natural heterogénea heterocigota, apta para favorecer el aumento de la base genética de las poblaciones cultivadas de palma de aceite.
Palabras clave: Elaeis guineensis Jacq., marcadores microsatélite, alelos, flujo genético.
Abstract
Oil palm Elaeis guineesis Jacq. is of great importance because of the oil extracted from its fruits, whose refined derivatives are commercially valuable as biodiesel, among others uses. This study sought to determine the structure and genetic diversity of 311 oil palm samples from the Republic of Cameroon with 10 microsatellite markers. The results showed values
for the average number of alleles per locus of Na= 8.433 and effective number of alleles per locus of Ne= 4.756; from the
differences between these values, it can be inferred that the 106 alleles found for these populations could be considered rare alleles. Additionally the value of genetic diversity was high (mean value of He= 0.781) compared to reports of several
authors. The obtained molecular variance showed that the highest percentage (80 %) was found within the individuals. The
analysis did not show any defined population structure, which allowed us to infer a high gene flow among the geographic
zones, corroborated it by the high genetic diversity values obtained. The 311 genotypes were defined as a heterogeneous
heterozygous natural population suitable to increase the genetic base of oil palm cultivated populations.
Key words: Elaeis guineensis Jacq, microsatellite markers, alleles, gene flow.
Introducción
La palma de aceite africana (Elaeis guineensis Jacq.) es una especie perenne originaria de África Central y Occidental; su cultivo es propio de las tierras bajas húmedas del trópico. La palma de aceite africana posee gran importancia comercial gracias al aceite que se extrae del mesocarpio, ya que de este tejido se obtiene el aceite para generar los productos para la industria alimenticia. Por otra parte se extraen otros ácidos grasos que están presentes en el endospermo de sus frutos, que también son importantes para la industria, por ejemplo para obtener la harina de coquito. Adicionalmente el aceite de palma, está siendo utilizado en la obtención de biodie sel a través de una reacción de trans-esterificación (FE- DEPALMA, 2009). Gracias al crecimiento de la industria, los programas de mejoramiento de la palma han cobra do importancia como una alternativa para contribuir al aumento de la rentabilidad de las plantaciones, mejorar la características como la calidad del aceite o la introducción de resistencia tanto a plagas como a enfermedades (Corley y Tinker, 2003).
La historia muestra que el material comercial sembrado mundialmente proviene de cuatro plantas cultivadas en el Jardín Botánico de Bogor de Java (Indonesia) en 1884, hecho que ha provocado uniformidad en el material productivo, generándose un efecto de estrechez en la base genética (Bakoumé et al., 2007; Cochard et al., 2009). Esto ha provocado la homogenización del cultivo, evidenciando la necesidad de ampliar la base genética de la palma de aceite, garantizando la obtención y conservación de una amplia gama de recursos genéticos, lo cual justifica la exploración y estudio de diversidad genética de materiales de palma provenientes del continente africano, en especial de Camerún, uno de los países centro de origen de esta especie. El Centro de investigación en palma de aceite (Cenipalma) hacia mediados del año 2007 con la cooperación científica del Institute for Agricultural Research and Development (IARD) de Camerún y el Ministerio de Agricultura y Desarrollo Rural (MADR), llevó a cabo la prospección y colecta de accesiones silvestres de palma de aceite en 6 zonas geográficas de la república de Camerún, estos materiales fueron sembrados en el Campo Experimental Palmar de la Vizcaína (Arias et al., 2011). Una de las herramientas utilizadas en el proceso de mejoramiento vegetal es el uso de marcadores moleculares, los cuales permiten dilucidar la diversidad genética de materiales silvestres, con el fin de acelerar la selección de individuos que van a ser incorporados en etapas de pre-mejoramiento.
La diversidad genética de una especie se puede analizar mediante el uso de marcadores moleculares, los cuales se definen como secuencias dentro de un genoma que permiten detectar las diferencias genotípicas (Mayes et al., 2000). Diversas técnicas de biología molecular se encuentran disponibles para la detección de variabilidad genética, entre los cuales se encuentran los basados en electroforesis de proteínas (Aloenzimas), hibridación de sondas "Southern Blot", RFLP (Restriction Fragmenth Length Polymorphism), minisatélites o VNTR (Variable Number of Tandem Repeats) y los basados en PCR como PCR-RFLP, RAPD (Random Amplified Polymorphic DNA), AFLP (Amplified Fragment Length Polymorphism) y los microsatélites que son Secuencias Simples Repetidas, conocidos como SSR. Estos últimos son los marcadores más utilizados para estudios de estructura y diversidad genética ya que presentan secuencias altamente conservadas (Rocha y Gómez, 2003), son codominantes, presentan multialelismo, y su distribución es al azar dentro del genoma. Algunos de los más importantes estudios dentro del género Elaeis son los realizados por Billotte et al. (2001), Bokumé et al. (2007) y Cochard et al. (2009) entre otros, quienes proyectan sus resultados en la aplicabilidad al interior de los programas de fitomejoramiento de esta especie. En este contexto, los estudios de diversidad genética de poblaciones naturales, mediante la utilización de técnicas moleculares, representan una herramienta útil en los programa de mejoramiento genético, en la medida que se involucran materiales no explotados comercialmente y con posible potencial agronómico. Así, la finalidad de este proyecto fue determinar tanto la estructura como la diversidad genética de materiales de palma de aceite provenientes de Camerún, resaltando su importancia como centro de origen.
Materiales y Métodos
Material vegetal
El material vegetal utilizado consistió de 311 genotipos de palma de aceite Elaeis guineensis (en estado de vivero), colectado en seis zonas geográficas de la República de Camerún (figura 1). Actualmente estas accesiones se encuentran sembradas en el Centro Experimental Palmar de la Vizcaína (Barrancabermeja- Santander) y hacen parte de la colección ex situ de Cenipalma. La estrategia de prospección de las palmas colectadas en Camerún, fue llevada a cabo inicialmente mediante un recorrido de 9700 km por tierra, para la identificación y selección de regiones geográficas con base a las condiciones climáticas, vegetación, suelos y altitud. En las zonas escogidas se tomaron muestras de racimos maduros, los cuales fueron organizados de acuerdo con las poblaciones espontáneas encontradas. Cada población espontánea fue catalogada como población independiente, si se delimitaba y se verificaba que por lo menos a más de 100 metros no hubiera más palmas. Posteriormente dentro de cada área se colecto un racimo por palma, al cual se le realizó una caracterización morfo-agronómica (Arias et al., 2011).
Extracción y cuantificación del ADN
El tejido foliar colectado en cada uno de los 311 genotipos, fue triturado utilizando nitrógeno líquido y posteriormente la extracción del ADN se realizó utilizando el kit DNeasy Plant Mini kit (250) ref: 69106 de QIAGEN, según las indicaciones del fabricante. El proceso para verificar la calidad y estimar la cantidad del ADN obtenido se realizó mediante dos metodologías; la primera fue la visualización de la muestra en gel de agarosa y su cuantificación por comparación de la fluorescencia de las bandas, observadas en el Foto documentador BioRad- Universal Hood II-. El segundo método utilizado, fue un procedimiento de espectro fotometría UV-visible a través de un espectrofotómetro JENWAY 6405 Uv/vis, utilizando una longitud de onda de 260 nm. Para verificar la calidad del ADN se observó la relación entre la longitud de onda de 260nm y 280nm. Las lecturas obtenidas por espectrofotometría se usaron para calcular la concentración de ADN obtenida para cada muestra y a partir de estas se realizaron las diluciones para uniformar la concentración de ADN de cada muestra a 5 ng/|jL
Amplificación SSR
La reacción de amplificación de SSR contenía una concentración final de 25 ng/jl de ADN, empleando 10 marcadores microsatélites reportados por Billotte et al. (2001), Billotte et al. (2005) y Singh et al. (2008). La amplificación de microsatélites mediante PCR se llevó a cabo en un volumen final de 15|l que contenía una solución amortiguadora de PCR 1X (20mM de Tris HCl, pH8.4 y 50 mM de KCl); 1.5 mM de MgCb, 0.2|M de cada dNTP y 1U de Taq DNA polimerasa (Invitrogen Life Technologies). Las condiciones de amplificación fueron: 1 minuto a 95°C (desnaturalización inicial), seguido por 35 ciclos de amplificación, cada uno compuesto por intervalos de temperatura a 94 °C durante 2 min, 52 °C por 30 seg, y 72 °C por 90 seg. Un paso de extensión final durante 8 min a 72°C. La verificación de los productos amplificados, se realizó mediante electroforesis en gel denaturalizante de poliacrilamida al 6 % y 5M de urea. Posteriormente, el gel fue teñido con nitrato de plata.
Análisis de datos
Para cada locus se determinó el número de alelos amplificados, utilizando como referencia los patrones de amplificación reportados por los autores. Para estimar la diversidad genética se tuvieron en cuenta los siguientes parámetros: frecuencia alélica (f), Número medio de alelos por locus (Na), número efectivo de alelos por locus (Ne), heterocigosidad observada (Ho), heterocigosidad esperada (He) (parámetro utilizado para cuantificar la diversidad genética) bajo el índice de Nei (1978), porcentaje de loci polimórficos (%P) e Indice de fijación (F). Para obtener esta información se hizo uso de los algoritmos incluidos en el programa GenALEX 6.1 (Peakall y Smouse, 2006). La estructura y diferenciación genética entre zonas se calculó mediante los estadísticos F (FST,FIS y FIT) de diferenciación de Wright (1965) y el Flujo genético (Nm) (Nei, 1987), fue estimado indirectamente a partir de FST (Wright, 1965) utilizando el programa GenALEX 6.1 (Peakall y Smouse, 2006). Para evaluar la estructura poblacional se realizó un Análisis de la Varianza Molecular (AMO- VA) con fuentes de variación entre y dentro de zonas geográficas. Con esta AMOVA se calcularon los estadísticos F. El test de significancia estadística se realizó a través de 999 permutaciones con el programa GenALEX 6.1 (Peakall y Smouse, 2006). Para conocer el tipo de relaciones genéticas entre zonas geográficas se realizó un Análisis de Coordenadas Principales, mediante el programa GenALEX 6.1 (Peakall y Smouse, 2006), utilizando la matriz de distancia genética de Nei (1972). Adicionalmente se realizó un Análisis de Coordenadas Principales, tomando como unidades los individuos y/o accesiones para el cual se utilizó la matriz de similitud genética de Nei y Li (1979); este análisis fue realizado mediante el programa NTSYS 2.1 (Rohlf, 2000).
Resultados y discusión
Polimorfismo
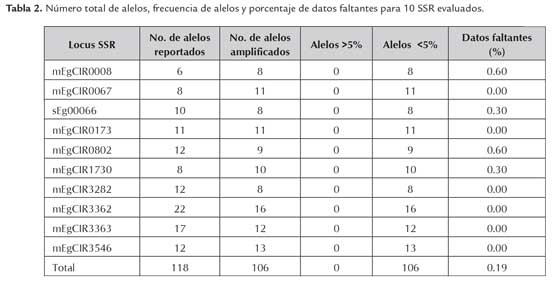
Los 10 SSR analizados presentaron un excelente resultado en la amplificación (figura 2), hecho que facilitó la obtención de la información. Para determinar la longitud de cada alelo y evitar la lectura de alelos falsos, se tuvieron en cuenta los rangos de longitud reportados por Billotte et al. (2001), Billotte et al. (2005), Bakoume et al. (2007) y Singh et al. (2008), pudiéndose verificar que los alelos observados en este trabajo se encontraron dentro de los rangos de amplificación reportados (tabla 1). Sin embargo los alelos observados para los locus sEg00066 y mEgCIR3282, se encuentran por encima del rango reportado por los autores. Lo anterior evidencia que aunque la longitud de los alelos reportados por otros autores, sirven como una guía para realizar una correcta lectura; no es una regla general. Debido a que el número de repeticiones de cada locus, así como el número de los alelos detectados depende de la naturaleza del germoplasma y por ende de su variabilidad.
El número de alelos obtenido por locus varió en un rango de 8 a 16, con un total de 106 alelos (tabla 2). En los loci mEgCIR0008 y mEgCIR0067 evaluados en este estudio se obtuvo un número mayor de alelos al reportado por Billote et al. (2001). Estos autores indican que para el locus mEgCIR0008 amplificaron 6 alelos y para el locus mEgCIR0067 8 alelos, en 18 muestras de palma de aceite E. guineensis de diferentes orígenes incluyendo Camerún. Para el marcador microsatélite sEg00066 se obtuvo un número menor de alelos al reportado por Sing et al. (2008), quienes indicaron que para este marcador amplificaron 10 alelos, en 76 muestras de E. guineensis de diferentes orígenes incluyendo Camerún. La cantidad de datos faltantes para los accesiones analizadas, alcanzó un porcentaje muy bajo (0.19%), en los 10 locus analizados para los 311 individuos, razón por la cual todos los marcadores fueron incluidos en el análisis de datos.
Al comparar el número de alelos obtenido en esta investigación con los reportados por Bakoumé et al. (2007), quienes evaluaron 494 muestras de E. guineensis de 10 países Africanos, los locimEgCIR1730 y mEgCIR3546 mostraron un número mayor de alelos; mEgCIR0173 evidenció el mismo número de alelos al reportado, mientras que para los demás loci el número de alelos fue menor al reportado. Los resultados anteriores mostraron que la cantidad de alelos está directamente relacionada con el número de muestras y con los diferentes orígenes utilizados en un estudio. Es importante reconocer que las 311 muestras de palma de aceite presentaron un alto número de alelos a pesar de incluirse un solo país de origen (Camerún), observación que concuerda con Bakoumé et al. (2007). Por otra parte las frecuencias reportadas en este estudio, mostraron que ninguno de los alelos encontrados para los loci analizados se presentó recurrentemente en porcentajes mayores al 5% (tabla 3). Al respecto, Ki mura (1983) y Joyce y Tavaré (1995) consideran que si la frecuencia de una alelo, medida bajo su frecuencia relativa, resulta menor que el valor establecido para q (0.5), como en este caso, los 106 alelos encontrados serán descritos como alelos raros.

Por otra parte la prueba de chi-cuadrada aplicada para todos los loci, excepto para el locus mEgCIR3282, fue altamente significativa (p < 0.001) (tabla 3). Por lo tanto se rechaza la hipótesis nula de que la distribución de las frecuencias es rectangular y se acepta la hipótesis alterna de que la distribución de las frecuencias no es rectangular o uniforme.
Diversidad genética dentro de cada zona geográfica
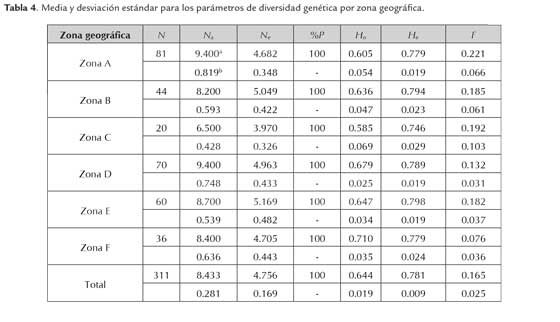
Como resultados relevantes se obtuvieron los valores promedio para el número medio de alelos por locus (Na=8.433) y el número efectivo de alelos por locus (Ne=4.756) (tabla 4). Laurentin (2009) indica que cuando estos valores son muy cercanos, las frecuencias alélicas son similares. Sin embargo, las amplias diferencias entre los valores de Na y Ne permiten inferir que los 106 alelos encontrados para estas poblaciones son considerados como alelos raros, confirmando los resultados obtenidos para el porcentaje de las frecuencias alélicas. Bakoumé et al. (2007) reportaron valores para el número medio de alelos por locus (Na) con rangos entre 4.4 a 5.5 y el número efectivo de alelos por locus (Ne) con rangos entre 2.5 a 3.9, para 102 materiales de palma de aceite de Camerún, valores que son menores a los reportados en esta investigación. El alto porcentaje de alelos raros encontrados podría estar relacionado con el porcentaje de loci polimórfico (%P), ya que todos los loci estudiados para las 6 zonas mostraron un 100% de polimorfismo bajo el criterio del 0,99. Es importante resaltar que la selección de los marcadores SSR fue influenciada por los reportes de los autores quienes diseñaron y describieron preliminarmente estos 10 cebadores como los más polimórficos dentro de sus investigaciones.
Por otra parte, la media de la heterocigosidad observada (Ho) para las 6 zonas estudiadas fue 0.644. La zona F presentó la Ho más alta (0.710) mientras que la zona C presentó la más baja (0.585). Bakoumé et al. (2007) reportaron un valor medio de Ho= 0.405 para 102 materiales procedentes de Camerún. Singh et al. (2008) reportaron un valor de Ho= 0.338 para 10 materiales procedentes de Camerún.
El valor de diversidad genética encontrado es similar entre las 6 zonas geográficas de Camerún, presentando valores de He que oscilan entre 0.746 y 0.798, con un valor medio de 0.781. Estos valores son mayores a los reportados por Billote et al. (2001) quienes presentaron un valor medio de He = 0.680 para 18 muestras de E. guineensis de diferentes orígenes, evaluadas con 21 SSR. Montoya et al. (2005) reportaron un valor de He= 0.454 para 48 muestras de palma de aceite provenientes de Angola evaluadas con 11 SSR. Bakoumé et al. (2007) reporta un valor de He = 0.660, para 102 materiales de Camerún y He = 0.644 para 494 muestras provenientes de 10 países Africanos evaluados con 16 SSR. Por otra parte Singh et al. (2008) reportaron para 10 muestras de palma de aceite provenientes de Camerún un valor de He=0.328 y una media de He= 0.356, para 76 muestras provenientes de países africanos evaluados con 10 EST (Expressed Sequence Tags)-SSR. Sin embargo no es posible realizar una comparación equitativa con nuestros resultados, puesto que por su naturaleza los marcadores EST-SSR, tienden a ser menos polimórficos, debido a que estos son obtenidos a partir de genes transcritos con una función putativa. Actualmente estos marcadores están siendo utilizados para la selección asistida por marcadores, especialmente cuando residen en los genes responsables de un rasgo fenotípico (Varshney et al., 2005). Caso contrario ocurre con los marcadores SSR obtenidos a partir del ADN genómico, que debido a esta característica son altamente polimórficos.
La diversidad genética en palma de aceite ha sido evaluada mediante otro tipo de marcadores moleculares como isoenzimas (Hayati et al., 2004 y Purba et al., 2000), RFLP (Maizura et al., 2006) y AFLP (Barcelos et al., 2002), que muestran valores de heterocigocidad esperada (He) para estas técnicas, generalmente menores a los valores de He obtenidos mediante marcadores tipo microsatélite. Por esto los marcadores SSR son reconocidos como una excelente y poderosa herramienta para estudios de diversidad genética (Weising et al, 2005 y Datta et al., 2009). El índice de fijación (F) obtenido corresponde a 0.165 para todas las poblaciones. Siendo que estos valores se miden en un rango que varía entre -1 que indica un alto grado de heterocigosidad y 1 que indica un alto grado de homocigosidad, se infiere el entrecruzamiento entre individuos, provocado un exceso de homocigotos. Estas desviaciones positivas pueden ser provocadas por tres factores esenciales: (1) altos niveles de endogamia, (2) cualidades de la estructura o subestructura poblacional, (3) el número limitado de individuos que conforman la población versus la cantidad de alelos obtenidos (Romero et al., 2003). Para este estudio el exceso de homocigotos podría estar asociado a las cualidades de la estructura o subestructura poblacional. Lo anterior ha sido sustentado por los valores de F (FIT, FST y FIS), los cuales revelaron un déficit de heterocigotos entre las zonas geográficas evaluadas (tabla 5).
Estructura Genética entre zonas geográficas
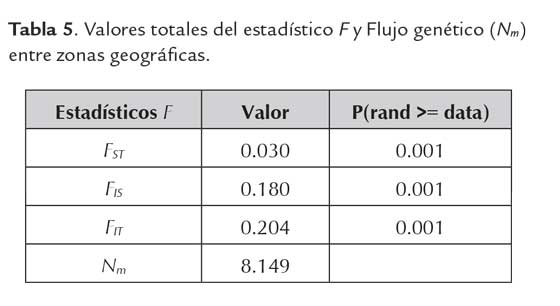
Los rangos de interpretación propuestos por Wright (1965), indicaron un bajo grado de diferenciación genética entre las 6 zonas geográficas, pues los valores obtenidos para el índice FST se encuentran distribuidos en un rango que oscila entre 0.013 a 0.049, los cuales son estadísticamente significativos. Los resultados del análisis de varianza molecular (AMOVA) muestran que el mayor porcentaje de la varianza (80%), se encuentra dentro de las zonas geográficas, mientras que el porcentaje restante se encuentra distribuido entre zonas geográficas (3%) y entre individuos (17%). Estos resultados son similares a los encontrados por Cochard et al. (2009) obteniendo como resultado que el 90.16 % de la varianza se encontraba distribuida dentro de orígenes. El término "origen" fue designado por estos autores, para referirse al sitio o zona geográfica en donde realizaron la colecta de las 318 accesiones analizadas con 14 marcadores SSR.
Los rangos de interpretación propuestos por Wright (1965) indicaron que el grado de diferenciación genética entre las 6 zonas es bajo, Fst= 0.030 (p > 0.001) (tabla 5), lo cual podría estar asociado con el alto número de migrantes entre poblaciones Nm = 8.093, evidenciando que existe recombinación genética entre las poblaciones evaluadas, sobrepasando los efectos de deriva e impidiendo diferenciación local (Slatkin, 1994). Por su parte los valores de Fit =0.204 (p > 0.001) y Fis=0.180 (p > 0.001) fueron positivos y significativos, indicando un déficit de heterocigosidad en la población como en cada sub población respectivamente, o la presencia de alelos nulos en la población que no pudieron ser detectados. De Vicente et al. (2004) explica que estos valores (Fst, Fis, Fit) tienden a modificarse en función de la cantidad de individuos estudiados, influyendo en la lectura real del flujo de genes que tiene lugar en la población.
Relaciones genéticas entre zonas geográficas
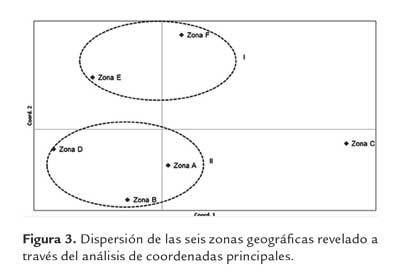
En el análisis de coordenadas principales realizado con la matriz de distancia genética de Nei (1972), mostró que las dos primeras coordenadas explican el 64.9% del total de la variación, la primera componente explica 34.47% y la segunda 30.45%. Dos grupos fueron visibles en las dos componentes (figura 3).
El grupo I está conformado por las zonas E y F. El grupo II esta con formado por tres zonas A, B y D. La zona C es la más distante a nivel genético. La matriz de la distancia genética de Nei (1972) fue interpretada con valores entre 0 (máxima igualdad genética) y 1 (máxima distancia genética). Según los valores obtenidos muestran que las zonas geográficas B y D son las más cercanas (0.0934), mientras que las zonas C y D fueron las más distantes (0.2441). De alguna forma lo anterior se ve reflejado en el mapa de localización de las zonas geográficas (figura 1), en donde las poblaciones B y D presentan la menor distancia geográfica entre ellas. La cercanía geográfica entre las zonas, facilita de cierta manera la dispersión de semilla, lo cual podría estar asociado con el alto número de migrantes (Nm = 8.149) encontrado en este estudio, que actúa como un efecto en la homogenización de la variación genética entre los grupos y sobrepasa los efectos de la deriva e impide la diferenciación local (Hedrick, 2005).
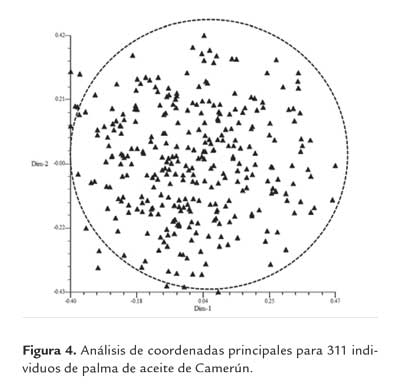
Por su parte los análisis de coordenadas principales tomando como unidades las 311 accesiones de palma de aceite, utilizando el coeficiente de similaridad genética de Nei & Li (1979) (figura 4) y el análisis de asignación de poblaciones (figura 5), mostraron a nivel espacial la conformación de un solo grupo; en donde no presentó ninguna asociación con los orígenes geo- gráficos en donde fueron colectadas las palmas.
Según estos resultados, se evidencia la ausencia de estructura poblacional definida, permitiendo inferir que existe un alto flujo genético entre los individuos, posiblemente sustentado por los altos valores de diversidad genética obtenidos. Epperson (2003) corrobora esta información revelando que la estructura genética puede ser difícil de identificar cuando aumenta la diversidad genética, o ser concreta cuando ésta disminuye. Además Slatkin (1994) y Neigel (1997), muestran que el flujo genético puede influenciar en la modificación de la distribución espacial de los genes, e intervenir en procesos como selección, recombinación y la mutación. Particularmente en la palma de aceite, el flujo genético podría estar directamente relacionado con el nivel de migración del polen (debida principalmente a dispersores aéreos) y a una activa dispersión de semillas, evitando la estructuración de la poblaciones y favoreciendo la diversidad genética de la especie (Ennos, 1994 y Corley y Tinker, 2003). Los resultados de esta investigación coinciden con Billote et al. (2001) y Montoya et al. (2005), quienes reportaron que las muestras analizadas no presentaron ningún tipo de estructura genética. Estos resultados han sido confirmados por Barcelos et al. (2002) quienes estudiando 38 accesiones de E. guineensis analizados a través de RFLP y 22 accesiones de palma de aceite analizados a través de AFLP, reportan un bajo nivel de estructuración genética dentro de todos los orígenes evaluados, lo que fue atribuido a la dispersión de material sin barreras geográficas en el continente africano. Maizura et al. (2006) a través de RFLP analizando 359 accesiones de palma de aceite provenientes de 11 países africanos encontró que las poblaciones incluidas en el estudio pudieron haber experimentado el flujo de genes entre las poblaciones dando lugar al alto nivel de heteroci- gosidad (He), atribuido a la dispersión de semillas a través de los ríos. En relación con estructura genética, los resultados de Hayati et al. (2004) contradicen lo reportado, indicando que el flujo genético obtenido (0.56) es bajo, denotando que de los 26 orígenes en los 10 países muestreados, la diferenciación genética entre las poblaciones de palma de aceite disminuyó a lo largo de una línea hacia el este de África, lo que indica que las poblaciones de África central y occiden- tal son más similares entre sí que con las poblaciones de África oriental. Sin embargo, es importante anotar que se incluyeron materiales de palma de aceite pro- venientes de la isla de Madagascar, lo cual pudo haber afectado la estimación del flujo genético y por ende la diferenciación genética.Conclusiones
Los diferentes análisis realizados en esta investigación evidenciaron la ausencia de estructura poblacional en las 311 muestras de palma de aceite E. guineensis pro- venientes de Camerún, infiriendo un alto flujo genéti- co entre los individuos, apoyado por los altos valores de diversidad genética encontrados. Estos resultados permiten referirse a los genotipos evaluados como una población natural heterogénea heterocigota, apta para favorecer el aumento de la base genética de las pobla- ciones cultivadas de palma de aceite.
Agradecimientos
Los autores expresan sus agradecimientos al Departa- mento Administrativo de Ciencia Tecnología e Inno- vación Colciencias, por su apoyo financiero para la realización de este trabajo bajo el Contrato No. 626- 2009. A Myriam Cristina Duque, Investigadora del Centro International de Agricultura Tropical (CIAT) y a Eloina Mesa, por la asesoría en el análisis de datos. Los autores también agradecemos a los doctores: Leo- nardo Rey y Claude Bakoumé, quienes realizaron la colecta de las accesiones de palma de aceite en Ca- merún. La investigación de Cenipalma es financiada por el Fondo de Fomento Palmero (FFP) administrado por Fedepalma.
Referencias bibliográficas
1. Arias, D.; Daza, E.; Montoya, C.; Romero, H. 2011.Colección genéti- ca de materiales de palma de aceite provenientes de Camerún. Rev. Palmas. 32(3): 27-36.
2. Bakoumé, C.; Wickneswari, R.; Raijanaidu, N.; Kushairi, A.; Amblard, P.; Billotte, N. 2007. Allelic diversity of natural oil palm (Elaeis guineensis Jacq.) populations detected by microsatellite markers. Implication in conservation. Rev. Palmas. 28(1): 149-158.
3. Barcelos, E.; Amblard, P.; Berthaud, J.; Seguin, M. 2002. Genetic- diversity and relationship in American and African oil palm as revealed by RFLP and AFLP molecular markers. Pesq. Agropec. Bras. 37(8):1105-1114.
4. Billotte, N.; Rusterucci, A.; Barcelos, E.; Noyer, J.; Amblard, P.; and Baurens, F. 2001. Development, characterization, and across- taxa utility of oil palm (Elaeis guineensis Jacq.) microsatellite markers. Genome. 44 (3): 413-425.
5. Billotte, N.; Marseillac, N.; Risterucci, A. M.; Adon, B.; Brottier, P.; Baurens, F. C.; ... & Charrier, A. 2005. Microsatellite-based high density linkage map in oil palm (Elaeis guineensis Jacq.). Theoretical and Applied Genetics. 110(4): 754-765.
6. Cochard, B.; Adon, B.; Rekima, S.; Billotte, N.; Desmier de Chenon, R.; Koutou, A.; Nouy, B.;, Omoré, A.; Purba, A.; Glazs- mann, J.; Noyer, J. 2009. Geographic and genetic structure of African oil palm diversity suggests new approaches to bree- ding. Tree genetics & genomes. 5:493-504
7. Corley, R.H.V.; and Tinker, PB. 2003. The oil palm. 4th Ed. World Agricultural Series. Blackwell Publishers Ltd, Oxford, UK.
8. Datta, S.; Kaashyap, M.; Kumar, S. 2009. Amplification of chickpea- specific SSR primers in Cajanus species and their validity in diversity analysis. Plant Breeding. 129(3): 334-340. doi:10.1111/ j.1439-0523.2009.01678.x.
9. De Vicente, M.; López, C.; Fulton, T. 2004. Genetic Diversity Analysis with Molecular Marker Data: Learning Module. International Plant Genetic Resources Institute (IPGRI).
10. Ennos, R. 1994. Estimating the relative rates of pollen and seed migration among plant populations. Heredity. 72: 250-259.
11. Epperson, B. 2003. Geographical Genetics. Princeton University Press: Englewood Cliffs, NJ.
12. Fedepalma. 2009. La Agroindustria de la palma de aceite en Colombia y el mundo. ISBN: 9789589834169.
13. Hayati, A.; Wickneswari, R.; Maizura, I.; Rajanaidu, N. 2004. Ge- netic diversity of oil palm (Elaeis guineensis Jacq) germplasm collections from Africa: implications from improvement and conservation of genetic resources. Theor. Appl. Genet. 108: 1274-1284.
14. Hedrick, P.W. 2005. Genetics of Populations (Third Edition). Jones and Bartlett, Boston. Joyce, P.; Tavaré, S. 1995. The distribution of rare alleles. J. Math. Biol. 33: 602-618.
15. Kimura, M. 1983. Rare variant alleles in the light of the neutral theory. Mol. Biol. Evol. 1: 84-93.
16. Laurentin, H. 2009. Data analysis for molecular characterization of plant genetic resources. Genetic Resources and Crop Evolution. 56(2): 277-292.
17. Maizura, I.; Rajanaidu, N; Zakri, A.; Cheah, S. 2006. Assessment of Genetic Diversity in Oil Palm (Elaeis guineensis Jacq) using res- triction fragment length polymorphism (RFLP). Genet. Resourc. Crop. Evol. 53(1):187-195(9).
18. Mayes, S.; Jack, P.L.; Corley, RHV. 2000. The use molecular markers to investigate the genetic structure of an oil palm breeding programme. Heredity. 85:288-293.
19. Montoya, C.; Arias, D.; Rey, L.; Rocha, P. 2005. Caracterización molecular de materiales de E. guineensis Jacq. Procedentes de Angola. Fitotecnia Colombiana. 5(2):1-10.
20. Nei, M. 1972. Genetic distance between populations. American Naturalist. 106: 283-292.
21. Nei, M. 1978. Estimation of average heterozigosity and genetic distance from a small number of individual. Genetics. 89:583-590.
22. Nei, M and Li, WH. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceedings of the National Academy of Science. 76: 5269-5273
23. Nei, M. 1987. Molecular evolutionary genetics. Columbia University Press.
24. Neigel, J. 1997. A comparison of alternative strategies for estimating gene flow from genetic markers. Annu. Rev. Ecol. Syst. 28:105-128.
25. Peakall, R.; Smouse, P. 2006. GenAlEx version 6.0: Genetic Analysis in Excel. Population genetic software for teaching and research. Molecular Ecology. 6: 288-295.
26. Purba, A.; Glazmann, J.; Noye, J. 2000. Geographic and genetic structure of African oil palm diversity suggests new approaches to breeding. Doi: 10.1007/s11295-009-0203-3.
27. Rocha, P.; Gomez, P. 2003. Marcadores moleculares, una herramienta útil para la selección genética de palma de aceite. Pal- mas. 24(2):9-25.
28. Rohlf, F. 2000. NTSYS pc: Numerical taxonomy and multivariate system, ver. 2.1, computer program. Setakuet, New York: Exeter Publishing.
29. Singh, R.; Zaki, N.; Ting, N.; Rosli, R.; Tan, S.; Low, E.; Maizura, I.; Cheah, S. 2008. Exploiting an oil palm EST database for the development of gene-derived SSR markers and their exploitation for assessment of genetic diversity Biologic. Biol. Cell. Mol. 63(2): 227-235.
30. Slatkin, M. 1994. Gene flow and population structure. Ecological Genetics, Editado por L. Real. pp. 3-17. Princeton University Press, Princeton, NJ.
31. Romero, C.; Pedryc, A.; Muñoz, V.; Llácer, G.; Badenes, M. 2003. Genetic diversity of different apricot geographical groups determined by SSR markers. Genome. 46: 244-252.
32. Varshney, K.R.; Graner, A.; Sorrells, M.E. 2005. Genomic microsate- llite markers in plants: features and applications. Trends in Biotechnology. 23(1): 48-55.
33. Weising, K.; Nybom, H.; Wolff, K.; Kahl, G. 2005. DNA Fingerprin- ting in Plants: Principles, Methods, and Applications, Second Edition. Taylor & Francis Group. United Estates.
34. Wright, S. 1965. The interpretation of population structure by F- statistics with special regard to system of mating. Evolution. 19: 395-420.
Referencias
Arias, D.; Daza, E.; Montoya, C.; Romero, H. 2011.Colección genéti- ca de materiales de palma de aceite provenientes de Camerún. Rev. Palmas. 32(3): 27-36.
Bakoumé, C.; Wickneswari, R.; Raijanaidu, N.; Kushairi, A.; Amblard, P.; Billotte, N. 2007. Allelic diversity of natural oil palm (Elaeis guineensis Jacq.) populations detected by microsatellite markers. Implication in conservation. Rev. Palmas. 28(1): 149-158.
Barcelos, E.; Amblard, P.; Berthaud, J.; Seguin, M. 2002. Genetic- diversity and relationship in American and African oil palm as revealed by RFLP and AFLP molecular markers. Pesq. Agropec. Bras. 37(8):1105-1114.
Billotte, N.; Rusterucci, A.; Barcelos, E.; Noyer, J.; Amblard, P.; and Baurens, F. 2001. Development, characterization, and across- taxa utility of oil palm (Elaeis guineensis Jacq.) microsatellite markers. Genome. 44 (3): 413-425.
Billotte, N.; Marseillac, N.; Risterucci, A. M.; Adon, B.; Brottier, P.; Baurens, F. C.; ... & Charrier, A. 2005. Microsatellite-based high density linkage map in oil palm (Elaeis guineensis Jacq.). Theoretical and Applied Genetics. 110(4): 754-765.
Cochard, B.; Adon, B.; Rekima, S.; Billotte, N.; Desmier de Chenon, R.; Koutou, A.; Nouy, B.;, Omoré, A.; Purba, A.; Glazs- mann, J.; Noyer, J. 2009. Geographic and genetic structure of African oil palm diversity suggests new approaches to bree- ding. Tree genetics & genomes. 5:493-504
Corley, R.H.V.; and Tinker, PB. 2003. The oil palm. 4th Ed. World Agricultural Series. Blackwell Publishers Ltd, Oxford, UK.
Datta, S.; Kaashyap, M.; Kumar, S. 2009. Amplification of chickpea- specific SSR primers in Cajanus species and their validity in diversity analysis. Plant Breeding. 129(3): 334-340. doi:10.1111/j.1439-0523.2009.01678.x.
De Vicente, M.; López, C.; Fulton, T. 2004. Genetic Diversity Analysis with Molecular Marker Data: Learning Module. International Plant Genetic Resources Institute (IPGRI).
Ennos, R. 1994. Estimating the relative rates of pollen and seed migration among plant populations. Heredity. 72: 250-259.
Epperson, B. 2003. Geographical Genetics. Princeton University Press: Englewood Cliffs, NJ.
Fedepalma. 2009. La Agroindustria de la palma de aceite en Colombia y el mundo. ISBN: 9789589834169.
Hayati, A.; Wickneswari, R.; Maizura, I.; Rajanaidu, N. 2004. Ge- netic diversity of oil palm (Elaeis guineensis Jacq) germplasm collections from Africa: implications from improvement and conservation of genetic resources. Theor. Appl. Genet. 108: 1274-1284.
Hedrick, P.W. 2005. Genetics of Populations (Third Edition). Jones and Bartlett, Boston. Joyce, P.; Tavaré, S. 1995. The distribution of rare alleles. J. Math. Biol. 33: 602-618.
Kimura, M. 1983. Rare variant alleles in the light of the neutral theory. Mol. Biol. Evol. 1: 84-93.
Laurentin, H. 2009. Data analysis for molecular characterization of plant genetic resources. Genetic Resources and Crop Evolution. 56(2): 277-292.
Maizura, I.; Rajanaidu, N; Zakri, A.; Cheah, S. 2006. Assessment of Genetic Diversity in Oil Palm (Elaeis guineensis Jacq) using res- triction fragment length polymorphism (RFLP). Genet. Resourc. Crop. Evol. 53(1):187-195(9).
Mayes, S.; Jack, P.L.; Corley, RHV. 2000. The use molecular markers to investigate the genetic structure of an oil palm breeding programme. Heredity. 85:288-293.
Montoya, C.; Arias, D.; Rey, L.; Rocha, P. 2005. Caracterización molecular de materiales de E. guineensis Jacq. Procedentes de Angola. Fitotecnia Colombiana. 5(2):1-10.
Nei, M. 1972. Genetic distance between populations. American Naturalist. 106: 283-292.
Nei, M. 1978. Estimation of average heterozigosity and genetic distance from a small number of individual. Genetics. 89:583-590.
Nei, M and Li, WH. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceedings of the National Academy of Science. 76: 5269-5273
Nei, M. 1987. Molecular evolutionary genetics. Columbia University Press.
Neigel, J. 1997. A comparison of alternative strategies for estimating gene flow from genetic markers. Annu. Rev. Ecol. Syst. 28:105-128.
Peakall, R.; Smouse, P. 2006. GenAlEx version 6.0: Genetic Analysis in Excel. Population genetic software for teaching and research. Molecular Ecology. 6: 288-295.
Purba, A.; Glazmann, J.; Noye, J. 2000. Geographic and genetic structure of African oil palm diversity suggests new approaches to breeding. Doi: 10.1007/s11295-009-0203-3.
Rocha, P.; Gomez, P. 2003. Marcadores moleculares, una herramienta útil para la selección genética de palma de aceite. Pal- mas. 24(2):9-25.
Rohlf, F. 2000. NTSYS pc: Numerical taxonomy and multivariate system, ver. 2.1, computer program. Setakuet, New York: Exeter Publishing.
Singh, R.; Zaki, N.; Ting, N.; Rosli, R.; Tan, S.; Low, E.; Maizura, I.; Cheah, S. 2008. Exploiting an oil palm EST database for the development of gene-derived SSR markers and their exploitation for assessment of genetic diversity Biologic. Biol. Cell. Mol. 63(2): 227-235.
Slatkin, M. 1994. Gene flow and population structure. Ecological Genetics, Editado por L. Real. pp. 3-17. Princeton University Press, Princeton, NJ.
Romero, C.; Pedryc, A.; Muñoz, V.; Llácer, G.; Badenes, M. 2003. Genetic diversity of different apricot geographical groups determined by SSR markers. Genome. 46: 244-252.
Varshney, K.R.; Graner, A.; Sorrells, M.E. 2005. Genomic microsate- llite markers in plants: features and applications. Trends in Biotechnology. 23(1): 48-55.
Weising, K.; Nybom, H.; Wolff, K.; Kahl, G. 2005. DNA Fingerprin- ting in Plants: Principles, Methods, and Applications, Second Edition. Taylor & Francis Group. United Estates.
Wright, S. 1965. The interpretation of population structure by F- statistics with special regard to system of mating. Evolution. 19: 395-420.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
CrossRef Cited-by
1. Sochinwechi Nwosisi, Kripa Dhakal, Dilip Nandwani, Joshua Ibukun Raji, Sarada Krishnan, Yoel Beovides-García. (2019). Genetic Diversity in Horticultural Plants. Sustainable Development and Biodiversity. 22, p.87. https://doi.org/10.1007/978-3-319-96454-6_4.
2. Christian Camilo Castañeda Cardona, Yacenia Morillo Coronado, Ana Cruz Morillo Conronado, Iván Ochoa. (2018). Genetic diversity in oil palm (Elaeis guineensis Jacq) using RAM (Random Amplified Microsatellites). Bragantia, 77(4), p.546. https://doi.org/10.1590/1678-4499.2017385.
3. Manuel Alejandro Sánchez. (2017). Estudio de la variabilidad genética en accesiones de papa (Solanum tuberosum L.) mediante marcadores SSRs. Ciencia y Agricultura, 14(2), p.67. https://doi.org/10.19053/01228420.v14.n2.2017.7150.
4. Elías Rangel L., Gloria Acened Puentes M., Luis Felipe Rodríguez C.. (2014). An analysis at the technology level of the oil palm (Elaeis guineensis Jacq.) plantations in the municipality of Tibu (Colombia). Agronomía Colombiana, 32(3), p.432. https://doi.org/10.15446/agron.colomb.v32n3.45931.
Dimensions
PlumX
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2014 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).