Distribución y diversidad genética de Begomovirus que infectan tomate (Solanum lycopersicum L) en Colombia
Palabras clave:
Geminivirus, Solanum licopersicum, PCR-RFLP, infecciones mixtas, mixed infections (es)Título en ingles: Distribution and genetic diversity of tomato –infecting Begomovirus in Colombia
Resumen: Las enfermedades causadas por los begomovirus, (Familia Geminiviridae) constituyen una serie limitante para la producción del tomate en Colombia. Sin embargo, la caracterización de estos virus no ha sido realizada al momento. Aquí presentamos los resultados de un muestreo a nivel nacional que buscaba conocer la distribución y diversidad genética de los geminivirus que están afectando el cultivo de tomate en Colombia. Los virus fueron detectados mediante PCR, empleando primer universales específicos para el género Begomovirus. Los fragmentos amplificados por PCR fueron sometidos a un análisis tipo RFLP cuyos resultados evidenciaron presencia de infecciones mixtas e individuales de geminivirus en la mayoría de las muestras recolectadas en campo. Los fragmentos amplificados por PCR fueron clonados y secuenciados. El análisis de secuencia y filogenético mostró que los aislados begomovirales colombianos eran gemininivirus bipartitas típicos del Hemisferio Occidental y que algunos eran variantes de PYMV y otros de ToTEV. Mediante el análisis de los elementos cis-regulatorios (iterones) presentes en el promotor del gen de la proteína asociada a replicación (Rep) de los begomovirus aislados es posible postular eventos de pseudorecombinación que podrían suceder entre ellos durante la ocurrencia de infecciones mixtas en tomate.
Palabras claves: Geminivirus; Solanum licopersicum; PCR-RFLP; infecciones mixtas.
Abstract: Diseased caused by begomoviruses (Family Geminiviridae) constitute a serious constraint to tomato production in Colombia. However characterization of these new viruses had not been carried out so far. Here we report a large scale survey on the distribution and genetic diversity of tomato infecting geminiviruses which are affecting mayor growing area of this crop in the country. Viruses were detected by PCR using universal primers for members of genus Begomovirus. The RFLP analysis of PCR-amplified fragments showed individual and mixed infections of several geminiviruses in many of the samples. PCR-amplified fragments were cloned and sequenced. Based on sequence comparations and phylogenetic analysis, the Colombian geminivirus isolates were new world bipartite geminiviruses showing close relationship with PYMV and ToVEV. By means of bioinformatic analysis of cis-acting elements (iterons) involved in DNA replication of rep gene of Colombian geminivirus isolates was possible to postulate possible pseudorecombinación events that could occur between them but also confirm the occurrence of mixed infections.
Keywords: Geminivirus; Solanum licopersicum; PCR-RFLP; mixed infections
ARTÍCULO DE INVESTIGACIÓN
Distribución y diversidad genética deBegomovirus que infectan tomate (Solanum lycopersicum L) en Colombia
Distribution and genetic diversity of tomato -infectingBegomovirus in Colombia
Juan Carlos Vaca-Vaca (MSc-PhD)1, Jhon Fredy Betancur-Pérez2, Karina López-López (Ph.D)3.
1 Ph.D, Biotecnología de Plantas; MSc. Microbiología con Énfasis en Biotecnología; Profesor del Departamento de Ciencias Agrícolas. Universidad Nacional de Colombia. AA 237. Palmira Valle del Cauca, Colombia. Autor para correspondencia:jcvacav@unal.edu.co
2 Candidato a Doctor en Ciencias Agropecuarias área de énfasis mejoramiento genético.
3 Ph.D, Biotecnología de Plantas; Profesora Asociada del Departamento de Ciencias Biológicas.
Recibido: noviembre 29 de 2011 Aprobado: junio 14 de 2012
Resumen
Las enfermedades causadas por los begomovirus, (Familia Geminiviridae) constituyen una serie limitante para la producción del tomate en Colombia. Sin embargo, la caracterización de estos virus no ha sido realizada al momento. Aquí presentamos los resultados de un muestreo a nivel nacional que buscaba conocer la distribución y diversidad genética de los geminivirus que están afectando el cultivo de tomate en Colombia. Los virus fueron detectados mediante PCR, empleando primer universales específicos para el género Begomovirus. Los fragmentos amplificados por PCR fueron sometidos a un análisis tipo RFLP cuyos resultados evidenciaron presencia de infecciones mixtas e individuales de geminivirus en la mayoría de las muestras recolectadas en campo. Los fragmentos amplificados por PCR fueron clonados y secuenciados. El análisis de secuencia y filogenético mostró que los aislados begomovirales colombianos eran gemininivirus bipartitas típicos del Hemisferio Occidental y que algunos eran variantes de PYMV y otros de ToTEV. Mediante el análisis de los elementos cis-regulatorios (iterones) presentes en el promotor del gen de la proteína asociada a replicación (Rep) de los begomovirus aislados es posible postular eventos de pseudorecombinación que podrían suceder entre ellos durante la ocurrencia de infecciones mixtas en tomate.
Palabras clave: Geminivirus, Solanum licopersicum, PCR-RFLP, infecciones mixtas.
Abstract
Diseased caused by begomoviruses (Family Geminiviridae) constitute a serious constraint to tomato production in Colombia. However characterization of these new viruses had not been carried out so far. Here we report a large scale survey on the distribution and genetic diversity of tomato infecting geminiviruses which are affecting mayor growing area of this crop in the country. Viruses were detected by PCR using universal primers for members of genus Begomovirus. The RFLP analysis of PCR-amplified fragments showed individual and mixed infections of several geminiviruses in many of the samples. PCR-amplified fragments were cloned and sequenced. Based on sequence comparations and phylogenetic analysis, the Colombian geminivirus isolates were new world bipartite geminiviruses showing close relationship with PYMV and ToVEV. By means of bioinformatic analysis of cis-acting elements (iterons) involved in DNA replication of rep gene of Colombian geminivirus isolates was possible to postulate possible pseudorecombinación events that could occur between them but also confirm the occurrence of mixed infections.
Key words: Geminivirus, Solanum licopersicum, PCR-RFLP, mixed infections.
Introducción
En Colombia, las investigaciones sobre enfermedades virales que afectan el cultivo de tomate han sido realizadas por medio de técnicas microscópicas, serológicas y/o moleculares, identificando a la fecha virus pertenecientes a los géneros: Begomovirus, (Tomato yellow mosaic virus -ToYMV), Tobamovirus (Tobacco mosaic virus -TMV y/oTomato mosaic virus -ToMV), Cucumovirus (Cucumber mosaic virus-CMV), Tospovirus (Tomato spotted wilt virus -TSWV, Impatients necrotic spot virus -INSV), Crinivirus (Potato yellow vein virus -PYVV), Potyvirus(Pepper deforming mosaic virus -PepDMV ) y Nepovirus, (Tobacco ringspot virus -TRSV) (Tamayo y Jaramillo, 2006; Uribe-Echeverri, et al. 2011).
Los virus clasificados dentro del género Begomovirus (Familia Geminiviridae) se caracterizan por tener uno o dos componentes de DNA cadena sencilla, el vector biológico es la mosca blanca (Bemisia tabaci Genn; Homoptera: Aleyrodidae) e infectan plantas dicotiledóneas (Rojas et al. 2005). Los begomovirus bipartitas tienen dos componentes genómicos, DNA-A y DNA-B. El DNA-A presenta cuatro marcos de lectura abiertos (MLA) los cuales codifican proteínas requeridas para la replicación, transcripción y encapsidación del virus. El DNA-B tiene dos MLA, los cuales codifican proteínas asociadas al movimiento viral y al desarrollo de síntomas (Hanley et al. 1999; Gutiérrez, 2000; Rojas et al. 2005). Rojas et al. (2005) describen que para el establecimiento de una infección sistémica eficiente se necesitan ambos componentes virales los cuales son encapsidados en forma independiente.
Los begomovirus han sido considerados como un grupo emergente de virus en plantas por su severidad en las enfermedades causadas. En las últimas tres décadas se ha observado una alta incidencia de estos virus en regiones tropicales y subtropicales del mundo, causando pérdidas devastadoras en producción en cultivos de frijol, yuca, algodón, cucurbitáceas y tomate (Morales y Anderson, 2001; Polston y Anderson, 1997). América Latina ha sido una región muy afectada en cuanto al surgimiento de nuevos begomovirus, número de cultivos afectados, pérdidas en las cosechas y áreas agrícolas devastadas (Morales y Anderson, 2001). Los síntomas típicos de la enfermedad causada por begomovirus son mosaicos amarillos brillantes, moteados amarillos, epinastias, clorosis foliar marginal, abultamientos foliares, reducción del área foliar, enanismo, retraso del crecimiento y reducción en tamaño de los frutos hasta la abscisión floral en casos extremos (Hanley et al. 1999; Gutiérrez, 2000; Rojas et al. 2005).
Reportes realizados por la FAO durante el periodo comprendido entre el año 1990 y el año 2007, estiman que la producción de tomate en Colombia pasó de 505.005 toneladas (en 23.400 ha) a 390.000 toneladas (en 15.000 ha), con una disminución en el área sembrada del 35.9% (FAOSTAT 2007). De acuerdo a lo reportado por Morales et al. (2002), una de las causas en la disminución en la producción del cultivo de tomate es la aparición de nuevas enfermedades virales, en especial aquellas causadas por virus del género Begomovirus.
Dada esta problemática begomoviral en cultivos de tomate, una estrategia para controlarlos es el uso de materiales de tomate resistentes a begomovirus endémicos del país. Un primer paso para desarrollar un programa de mejoramiento genético de tomate con resistencia a virus es conocer la identidad y diversidad genética de los begomovirus que están afectando el cultivo en Colombia, con el fin de buscar materiales resistentes al begomovirus que prevalecen en las zonas productoras de tomate. En el presente trabajo de investigación se reporta que las variantes begomovirales que actualmente están afectando el cultivo de tomate en Colombia están relacionados filogenéticamente con el Virus del mosaico amarillo de la papa (PYMV) y con el Tomato venezuela virus (ToVEV).
Materiales y métodos
Recolección del material vegetal
Se colectaron hojas jóvenes de plantas de tomate en 74 fincas en los departamentos del Valle del Cauca, Cundinamarca, Antioquia, Boyacá y Santander con síntomas característicos de la enfermedad begomoviral tales como: mosaicos amarillos brillantes, epinastias, clorosis foliar marginal, abultamientos foliares, reducción del área foliar, enanismo y retraso del crecimiento (Polston y Anderson, 1997). Los sitios de colecta fueron georeferenciados mediante el equipo GPS-CSX-60 (Garmin ®).
Extracción de DNA total
Con el propósito de llevar a cabo los diferentes análisis moleculares, el tejido foliar de cada muestra colectada fue macerado en nitrógeno líquido (N2) y usado para la extracción del DNA genómico, este proceso se llevó a cabo siguiendo las recomendaciones del fabricante DNeasy Plant mini Kit (Qiagen, Germany).
Reacción en Cadena de la Polimerasa (PCR)
Para la detección del begomovirus se utilizó la reacción en cadena de la polimerasa - PCR (Polymerase Chain Reaction), usando los iniciadores y las condiciones de PCR previamente reportadas por Rojas et al. (1993). La identificación del componente A se realizó usando los juegos de iniciadores PAL1v1978 / PAR1c496, los cuales amplifican una región de 1.1kb y la identificación del componente B con los iniciadores PBL1v2040 / PCRc1 los cuales amplifican una región de 0.6kb. El equipo que se usó para la amplificación fue un termociclador ICycler® de BioRad ®. Los productos amplificados fueron observados en geles de agarosa al 1% (p/v) teñidos con bromuro de etidio (10 ng/µl), visualizados en un transiluminador de luz ultravioleta (Molecular Imager Gel DocXR+ Systems BIORAD ®) y analizados mediante el uso del software Quantity One - 4.6.5 provisto por el fabricante del equipo.
PCR-RFLP
Para determinar polimorfismos entre los fragmentos amplificados del componente A begomoviral, se llevó a cabo la técnica molecular PCR-RFLP (Polymerase Chain Reaction - Restriction Fragment Length Polymorphism) (Sambrook y Russell, 2001), la cuál ha sido reportada para realizar estudios de diversidad genética de geminivirus (Willment et al. 2001). Para seleccionar las enzimas de restricción se realizó una búsqueda y análisis bioinformático en el Genebank (Benson et al. 2011) de secuencias reportadas para el componente genómico A de begomovirus que están afectando el cultivo de tomate en áreas del Caribe, Centro y Sur América, seleccionando las siguientes:Tomato yellow mosaic virus isolate Valle del Cauca (AY126610.1); Potato yellow mosaic Panamá virus (Y15034.1); Potato yellow mosaic Trinidad virus (AF039031.1); Potato yellow mosaic virus from Martinique (AY126610). Estas secuencias fueron procesadas en el programa Editseq® (software DNASTAR®) con el fin de contar con la secuencia parcial de aproximadamente 1100 pb (secuencia parcial de la proteína de la cápside, región común y proteína asociada a replicación) que representan los fragmentos que se amplificaron con los iniciadores de Rojas et al. (1993) utilizados para la reacción de PCR. Las secuencias parciales de los componentes genómicos A fueron cortados con diferentes enzimas de restricción en el programa MapDraw® (software DNASTAR®) (datos no mostrados). Con base en este análisis bioinformático se seleccionaron las enzimas de restricción HincII, PstI, RsaI y SspI como mejores candidatos para el análisis por RFLP (Tabla 2, restricción enzimática in silico).
Para la restricción enzimática in vivo, los fragmentos de 1100pb del componente A fueron amplificados por PCR y digeridos con cuatro enzimas de restricción: RsaI, PstI, SspI y HincII (Invitrogen ®). Las reacciones de digestión se llevaron a cabo siguiendo las metodologías descritas por el fabricante y los productos obtenidos fueron separados y visualizados en geles de poliacrilamida al 6% teñido con sales de plata.
Clonación y secuenciación
Los fragmentos de 1.1kb del componente A fueron purificados mediante el kit (Wizard® Genomic DNA Purification Promega ®) y clonados en el vector TOPO TA (Invitrogen®), siguiendo la metodología descrita por el fabricante. Los fragmentos de 0.6kb del componente B fueron purificados mediante el kit (Wizard Genomic DNA Purification Promega®), digeridos con la enzima PstI y clonados en el vector pUC19, utilizando la enzima T4 ligasa. Las reacciones de ligación fueron transformadas en células competentes Escherichia coli (One Shot, Invitrogen®) por el método de choque térmico (Sambrook y Russell 2001). A las colonias transformadas se les extrajo el DNA plasmídico con el kit QIAprep Spin Miniprep (Qiagen, Germany) siguiendo la metodología descrita por el fabricante. Para verificar la presencia del inserto se hizo un análisis de restricción usando las enzimas de restricción EcoRI y PstI, para liberar el inserto del vector TOPO TA y pUC19, respectivamente. Para la secuenciación de los fragmentos clonados se utilizaron los iniciadores M13 Forward y M13 Reverse, esta fue llevada a cabo por la empresa Macrogen, la cual emplea el método fluorescente del kit de secuenciación enzimática The Big Dye Terminador DNA (Perkin - Elmer Inc., Branchburg, NJ) y un secuenciador ABI Prisma 377 (Applied Biosystem Inc. Foster City, CA).
Análisis bioinformático
Las secuencias fueron editadas y ensambladas mediante el programa CodonCode Aligner® (CodonCode Corporation). Para determinar la identidad de los begomovirus hallados en la presente investigación, se realizó una comparación de secuencias mediante el algoritmo blastn (Zhang et al. 2000) disponible en la página web deNacional Center for Biotechnology Information (www.ncbi.nlm.nih.gov). La edición y alineamiento múltiple de las secuencias parciales correspondientes a 151 nt de la región 5' del Marco de Lectura Abierto (MLA) del gen de la proteína de la capside (CP) de los begomovirus aislados y otros reportados en la base de datos GenBank (Benson et al. 2011), se realizó en los programas Bioedit (Hall, 1999) y ClustalW (Thompson et al. 1997). A partir de estos datos se construyó una matriz de identidad de secuencias de nucleótidos en el programa BioEdit®.
Para establecer y verificar las relaciones de parentesco y geográficas entre el grupo de begomovirus identificados con otro grupo de begomovirus reportados en la base de datos GenBank GenBank (Benson et al. 2011), se realizaron análisis filogenéticos de las secuencias parciales correspondientes a 151 nt de la región 5' del Marco de Lectura Abierto (MLA) del gen de la proteína de la capside (Cp) en el programa Geneious 4.8.3 ® Biomatters Ltd (www.geneious.com); la similitud genética se calculó de acuerdo al modelo de distancia genética de Tamura-Nei (Nei, 1978) y el análisis de agrupamiento se realizó utilizando el método UPGMA para secuencias de nucleótidos (método gráfico de agrupamiento por parejas, que usa el promedio aritmético no ponderado).
La identificación de secuencia repetitivas (regiones regulatorias o iterones) localizados en la región promotora del gen de la proteína asociada a replicación (Rep) de los begomovirus fue realizado con el algoritmo ClustalW presente en la herramienta MEGA (Tamura et al. 2011). Los begomovirus fueron alineados utilizando como señales el codón de inicio de Rep, la caja TATA de Rep y los interones reportados para PYMV-Guadalupe (Ramos et al. 2003).
Las siguientes secuencias fueron tomadas de la base de datos GenBank (Benson et al. 2011) para realizar los análisis bioinformáticos:
Abutilon mosaic Bolivia virus (AbMBV, HM585445),
Abutilon mosaic virus (AbMV, X15983),
African cassava mosaic virus (ACMV, J02057),
Bean dwarf mosaic virus (BDMV, M88179),
Bean golden mosaic virus-[Brazil] (BGMV, M88686),
Bean golden yellow mosaic virus-[Puerto Rico] (BGYMV, M10070),
Chino del tomate virus_Sinaloa (CdTVTo, AF101476.1),
Leonurus mosaic virus (LeMV, U92532),
Malvastrum yellow mosaic virus (MalYMV, FJ600483.1)
Pepper golden mosaic virus (PepGMV, AY928516.1);
Pepper huasteco yellow vein virus_Mexico (PHYVV, X70418.1),
Potato yellow mosaic Panamá virus (PYMPV, Y15034.1),
Potato yellow mosaic Trinidad virus (PYMV-TT, AF039031.1),
Potato yellow mosaic virus from Martinique (PYMV- Ma, AY126610),
Potato yellow mosaic virus Puerto Rico (PYMV-To [PR:Tom:04], AY965897).
Potato yellow mosaic virus-[Guadalupe] (PYMV-To [GP:Tom], AY120882);
Potato yellow mosaic virus-[Venezuela] (PYMV-Po [VE], D00940),
Sida Brazil virus (SiBV, FN436001),
Sida golden mosaic Costa Rica virus (SGMCRV, X99550),
Sida golden mosaic virus (SiGMV, AF049336),
Squash leaf curl virus (SqLCV, M38183),
Tomato chlorotic mottle virus-[Brazil] (ToCMV, AF490004),
Tomato golden mosaic virus (TGMV, K02029),
Tomato leaf curl New Delhi virus (ToLCNDV, U15016),
Tomato leaf curl virus_Australia (ToLCV-To, S53251),
Tomato mosaic Barbados virus (ToMBV, AF213013),
Tomato mottle Taino virus (ToMoTV, AF012300.1),
Tomato mottle leaf curl Zulia virus (TMoLCV-Zulia, HQ171986.1)
Tomato mottle virus (ToMoV, L14460),
Tomato rugose mosaic virus [Brazil] (ToRMV, AF291705),
Tomato yellow leaf curl Sardinia virus [Italy] (TYLCSV, X61153),
Tomato yellow leaf curl virus-Mild [Portugal] (TYLCV, AF105975),
Tomato yellow mosaic virus isolate Valle del Cauca (PYMV-Valle, EU518935),
Tomato yellow spot virus from Argentina (ToYSV-Ar, FJ538207),
Tomato yellow vein streak virus [Argentina] (TYVSV-Ar, GQ387369),
Tomato yellow vein streak virus [Brazil] TYVSV-Br U79998),
Venezuela tomato geminivirus (ToVEV, AF026464),
Wissadula golden mosaic virus (WGMV, WGU69280).
Resultados y discusión
Evidencia de infecciones mixtas de begomovirus que afectan tomate en Colombia.
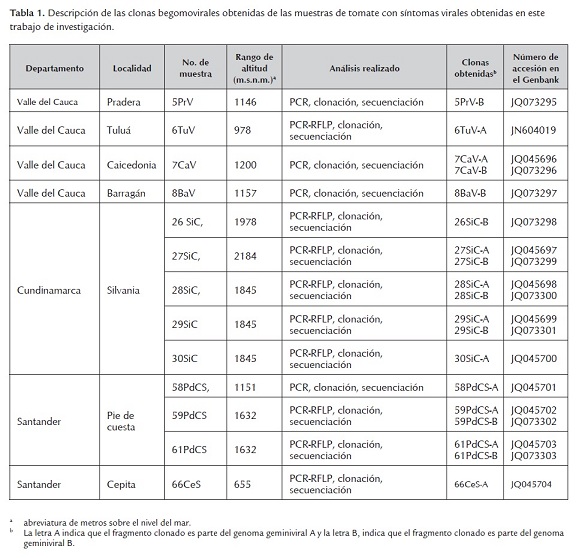
Conociendo la alta incidencia que han tenido los Begomovirus (Familia Geminiviridae) en zonas productoras de tomate de la región Andina Colombiana y el impacto que estos virus ha generado en los agricultores, se realizaron colectas de material vegetal (hojas de tomate) con síntomas virales característicos de begomovirus en los departamentos del Valle del Cauca, Boyacá, Cundinamarca, Antioquia y Santander (Vaca-Vaca et al. 2011). Para determinar la presencia de infecciones mixtas de begomovirus bipartitas en las muestras colectadas, se realizó una extracción de DNA total y mediante PCR se amplificaron dos fragmentos de 1100pb y 600pb, correspondientes a una sección del componente geminiviral A y B, respectivamente. Por medio de esta estrategia molecular se evidenciaron begomovirus bipartitas pertenecientes a la Familia Geminiviridae en 24 muestras provenientes de los departamentos de Valle del Cauca, Cundinamarca y Santander (tabla 1).
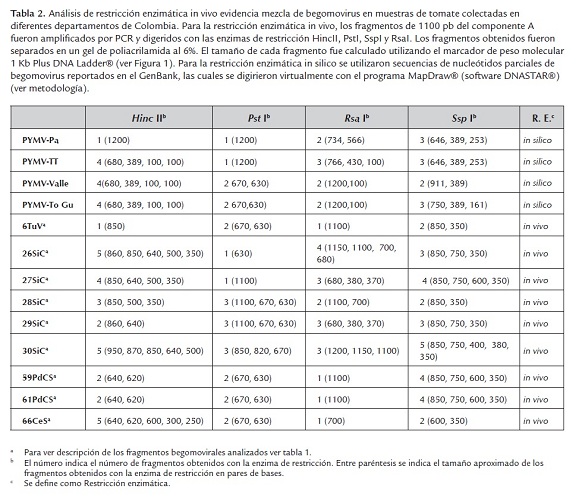
Con el fin de caracterizar las muestras positivas para begomovirus y determinar polimorfismos entre los diferentes virus detectados, se procedió a realizar la técnica de RFLP (Polimorfismo de Longitud de Fragmentos de Restricción) a los diferentes fragmentos amplificados por PCR utilizando las enzimas de restricción HincII, PstI, RsaI y SspI (tabla 2). Para realizar este análisis se emplearon 9 fragmentos de 1100 pb provenientes de Valle del Cauca (6TuV), Cundinamarca (26SiC, 27SiC, 28SiC, 29SiC, 30SiC) y Santander (59PdCS, 61PdCS y 66CeS). Los análisis de PCR-RFLP permitieron evidenciar la presencia de polimorfismos begomovirales entre las muestras de los diferentes departamentos (figura 1).
En la muestra del Valle del Cauca (figura 1, carril 1) se observó digestión del fragmento de 1100pb con las enzimas de restricción RsaI, PstI, SspI y HincII y la suma de los fragmentos obtenidos con las diferentes enzimas fue de 1100bp aproximadamente, lo que indicaba que en la muestra del Valle del Cauca predominaba una única variante begomoviral afectando los cultivos de tomate. En las muestras de Cundinamarca (figura 1, carril 2-6) se observó digestión con todas las enzimas de restricción utilizadas en el análisis mostrado un alto polimorfismo entre ellas. Con la enzima Pst I se obtuvieron tres patrones de restricción: a) un fragmento de 1100 pb para la muestras 27SiC, 28SiC y 29SiC; b) dos fragmentos de 670 pb y 630 pb para las muestras 26SiC, 28SiC, 29SiC y 30SiC, patrón que fue similar al encontrado en la muestra de Valle y muestras de Santander; c) dos fragmentos de 850 y 820pb en la muestra 30SiC. Los resultados de este análisis permitieron evidenciar la presencia de tres entidades begomovirales diferentes en las muestras recolectadas en el departamento de Cundinamarca y se encontró que en la muestras 28SiC, 29SiC y 30SiC, existían al menos dos entidades begomovirales presentes en cada muestra analizada. Este mismo resultado se observó cuando el análisis de restricción se realizó con la enzima Ssp I, donde la suma de los fragmentos liberados por algunas muestras (26SiC, 27SiC, 29SiC y 30SiC) es mayor de 1100 pb, evidenciando la presencia de más de un begomovirus por muestra analizada (figura 1, carril 2- 6). Para el caso de las muestras recolectadas en el departamento de Santander (figura 1, carril 7-9), el análisis tipo PCR-RFLP arrojó dos resultados: las muestras 59PdCS y 61PdCS presentaron dos patrones de restricción, implicando la presencia de al menos dos begomovirus diferentes; por su parte la muestra 66CeS presentó un único patrón de restricción. Este patrón de restricción fue similar al observado cuando se analizaron las muestras 59PdCS y 61PdCS por esta metodología. Por ejemplo, con la enzima Ssp I se liberan cuatro fragmentos en las muestras 59PdCS y 61PdCS de 850, 750, 600 y 350pb; mientras que la muestra 66CeS libera solo dos fragmentos de 600 y 350pb, respectivamente (figura 1, carril 9). Es importante mencionar que las muestras 59PdCS y 61PdCS mostraron el mismo patrón de restricción con las cuatro enzimas utilizadas (tabla 2), lo que indica que ambas muestras presentan infecciones mixtas causadas por los mismos virus.
Estos resultados evidenciaron una distribución geográfica de los begomovirus que están afectando tomate en Colombia: hay un único begomovirus afectando el Valle; al menos tres begomovirus afectando Cundinamarca mientras que para el caso de Santander, se observaron al menos dos geminivirus afectando esta hortaliza. El patrón de restricción presentado por el begomovirus de Valle del Cauca fue también observado en muestras analizadas de Cundinamarca y Santander, lo que estaría indicando que este aislado geminiviral, podría estar presente en las áreas productoras de tomate ubicadas en estos departamentos. Sin embargo, vale la pena anotar que algunos de los patrones de restricción observados en las muestras de Cundinamarca y Santander son exclusivos de cada una de estos departamentos. Estos resultados en conjunto nos indicarían que al menos cuatro aislados geminivirales diferentes están afectando cultivos de tomate en Colombia.
Otro resultado obtenido de este análisis tipo PCR-RFLP es la detección de mezclas de begomovirus afectando cultivos de tomate en áreas biogeográficas particulares de Colombia. Este fue el caso de las muestras colectadas en los departamentos de Cundinamarca y Santander en los cuáles se evidencio la presencia de mezclas de al menos dos geminivirus en cada muestra analizada. La presencia de infecciones mixtas de dos o más geminivirus infectando una misma planta son comunes en cultivos ubicados en zonas tropicales y subtropicales del planeta (Harrison et al. 1997; Sanz et al. 2000). Actualmente hay evidencias que indican que la aparición de enfermedades geminivirales cada vez más severas pueden ser debido a las interacciones que entre este tipo de virus se pueden verificar cuando están presentes en infecciones mixtas (Pita et al. 2001). Entre geminivirus que se encuentran coinfectando una misma planta (infección mixta) diferentes tipos de interacciones pueden verificarse entre ellos: algunas de ellas pueden implicar relaciones de sinergismo, que a la postre pueden conducir a la generación de enfermedades en el tomate con sintomatologías más acentuadas que aquellas producidas por cada virus por separado, o también pueden verificarse interacciones virales antagónicas en donde la presencia de un geminivirus particular interfiere con el desarrollo normal del ciclo infectivo del otro geminivirus presente en una misma planta conduciendo a infecciones cuyas sintomatologías son generalmente atenuadas (Méndez et al. 2003). Si cualquiera de estos tipos de interacción entre geminivirus se está verificando de manera particular en los cultivos de tomate de Santander y Cundinamarca es un tópico interesante de abordar en futuras investigaciones.
El cultivo de tomate en Colombia es afectado por begomovirus relacionados con el Potato yellow mosaic virus y el Tomato venezuela virus
Con el fin de establecer la identidad de los begomovirus previamente detectados por PCR-RFLP, se procedió a clonar y secuenciar los productos amplificados de los componentes A y B (tabla 1). Para el componente genómico geminiviral A, se obtuvieron 13 clones en total: dos del Valle del Cauca provenientes de Tuluá (6TuV-A) y Caicedonia (7CaV-A); Siete clones de Cundinamarca, provenientes de fincas ubicadas en la localidad de Silvania (27SiC-A, 28SiC-A, 29SiC1-A, 29SiC2-A, 30SiC1-A, 30SiC2-A, 30SiC3-A) y cuatro clones de Santander provenientes de las localidades de Pie de Cuesta (58PdCS-A, 59PdCS-A, 61PdCS-A) y Cepita (66CeS-A). Para el caso del componente genómico geminiviral B, se obtuvieron nueve clones en total: tres del Valle del Cauca provenientes de Pradera (5PrV-B), Caicedonia (7CaV-B) y Barragán (8BaV-B); cuatro de Cundinamarca provenientes de fincas en la localidad Silvania (26SiC-B, 27SiC-B, 28SiC-B, 29SiC-B) y dos de Santander provenientes de la localidad de Pie de Cuesta (59PdCS-B, 61PdCS-B).
Estos 22 fragmentos clonados fueron secuenciados, ensamblados y analizados en el programa blastn (Zhang et al. 2000). Los fragmentos correspondientes al componente genómico A presentaron un rango de longitud entre 1186 - 1287 bp y los fragmentos correspondientes al componente genómico B, una longitud de 510 - 558 bp. El análisis bioinformático de la secuencia de nucleótidos de los fragmentos 29SiC1-A y 29SiC2-A indicó la presencia del mismo fragmento clonado, por lo tanto a partir de este momento se denominó 29SiC-A. Este mismo hecho se observó para los fragmentos provenientes de la muestra 30SiC-A, los cuales mostraron ser idénticos entre sí. La secuencia de nucleótidos de estos 19 fragmentos fue depositada en la base de datos Genbank (tabla 1).
El análisis con blastn para cada una de las secuencias de nucleótidos provenientes del componente A geminiviral permitió determinar la presencia de 3 entidades virales relacionadas con el Virus del mosaico amarillo de la papa (Potato yellow mosaic virus- PYMV) y Tomato venezuela virus (ToVEV) (tabla 3). Dos fragmentos aislados de Valle del Cauca (6TuV-A, 7CaV-A) y dos de Cundinamarca (28SiC-A, 30SiC-A) presentaron su mayor porcentaje de identidad (96-99%) con Tomato yellow mosaic virus isolate Valle del Cauca (AY126610.1) (nombre propuesto no aceptado oficialmente por ICTV ya que se considera este puede ser una variante de PYMV; en este escrito se propone el acrónimo PYMV-Valle para referirse a este virus), un begomovirus previamente reportado en Colombia infectando cultivos de tomate en la localidad de Rozo- Valle del Cauca (Martínez et al. 2008). Dos clones identificados en Cundinamarca (27SiC-A y 29SiC-A) presentaron identidades de 83% con Venezuela tomato geminivirus (AF026464.1), un begomovirus aislado de cultivos de tomate en Venezuela (Guzmán et al. 1997) y posteriormente denominado ToVEV (Tomato Venezuela virus) por Argüello-Astorga & Ruiz-Medrano (2001). Por su parte, tres clones aislados en el departamento de Santander (58PdCS-A, 59PdCS-A, 61PdCS-A) presentaron valores de identidad de 93-94% con Potato yellow mosaic virus aislado Martinique (AF026464.1) un begomovirus reportado en el año 2004 infectando plantas de tomate en isla Martinica (Urbino et al. 2004), mientras que el clon 66CeS-A también aislado en el departamento de Santander presentó una identidad de 83% con Venezuela tomato virus (ToVEV). Estos resultados permiten afirmar que en las infecciones mixtas de geminivirus detectadas en este estudio afectando el cultivo tomate en Colombia hay tanto aislados begomovirales relacionados con PYMV como variantes lejanamente relacionados con el geminivirus ToVEV.
Para los fragmentos parciales del componente genómico geminiviral B, el análisis blastn de los clones 5PrV-B, 7CaV-B, 8BaV-B, 26SiC-B, 27SiC-B, 28SiC-B y 29SiC-B mostró valores de identidad del 81 al 92% con PYMV aislado Panamá (Y15033.1). Mientras que el clon 59PdCS-B presentó una identidad de 82% con PYMV aislado Puerto Rico (AY126613.1) y el clon 61PdCS-B una identidad de 82% con PYMV aislado Trinidad & Tobago (AF039032.1). La amplia diversidad a nivel de secuencia que presenta el fragmento correspondiente al componente genómico B con otros geminivirus reportados en el Genebank es una situación bastante común entre cepas de geminivirus bipartitas (Brown et al. 2005).
En conjunto estos resultados permiten plantear la posibilidad de que posibles eventos de pseurecombinación podrían ocurrir (Padidam et al. 1999) entre estos geminivirus, cuando éstos se encuentren presentes en infecciones mixtas en plantas de tomate. Si estos posibles eventos de pseudorecombinación se constituyen en una fuente de origen de nuevas variantes geminivirales que conduzcan al desarrollo de patologías más severas en este cultivo será materia de futuras investigaciones.
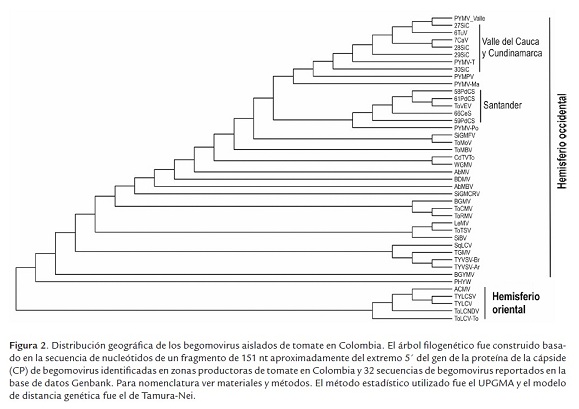
El gen de la cápside (CP) presenta el mayor grado de conservación de todos los genes de los geminivirus (Wyatt & Brown, 1996), por tanto a partir del análisis de la secuencia del gen CP se puede preliminarmente inferir la identidad de un begomovirus particular, así como su distribución geográfica y su relación con su vector biológico (Brown et al. 2001; Fauquet et al. 2008). A partir de este criterio se procedió a realizar una matriz de identidad (tabla 4) utilizando aproximadamente 151nt del extremo 5'del MLA de CP de cada uno de los 10 begomovirus aislados y se compararon contra las secuencias de un grupo de begomovirus reportados previamente afectando el tomate en otras latitudes del planeta y cuyas secuencias reposan en el GenBank (ver metodología). Por medio de este análisis se pudo evidenciar que dos begomovirus diferentes están afectando el tomate en Colombia: un primer grupo de ellos (clones 6TuV-A, 7CaV-A, 27SiC-A, 28SiC-A, 29SiC-A y 30SiC-A recolectados en los departamentos del Valle del Cauca y Cundinamarca), están relacionados con el PYMV tentativamente reportado como PYMV-Valle, con valores de identidad del 0,97 al 1. Este resultado podría indicar que PYMV emergió simultáneamente en estas dos regiones del país bien sea por la diseminación efectiva del mismo por parte de su vector biológico o bien sea por el transporte de material vegetal infectado por este virus de entre estas dos regiones. Un segundo grupo de geminivirus (clonas 58PdCS-A, 59PdCS-A, 61PdCS-A y 66CeS-A todos provenientes de muestras colectadas en el departamento de Santander) que se encontraron afectando el cultivo de tomate en Colombia están relacionados con Tomato venezuela virus (ToVEV) con valores de identidad del 0,96 - 0,99. Es posible que la prevalencia de PYMV así como de ToVEV afectando el cultivo de tomate en Colombia en diferentes zonas geográficas del país pueda ser el producto de una rápida emergencia, a lo largo de un periodo medianamente largo de adaptación a este hospedero, lo que condujo a un desplazamiento de cepas virales menos adaptadas al mismo, como se ha documentado en México y Brasil en los últimos años (Polston & Anderson, 1997; Riveiro et al. 2003). Este mismo resultado puede reflejarse en el árbol filogenético elaborado con las secuencias de CP de los geminivirus encontrados afectando el cultivo de tomate en campo (figura 2) en donde el porcentaje de identidad para miembros de un mismo grupo estaba en concordancia con el criterio de identidad de CP (Brown et al. 2001). Estos resultados indican que diferentes cepas de begomovirus están afectando el cultivo de tomate en Colombia en diferentes zonas biogeográficas del país: es decir que los geminivirus que afectan el tomate en los departamentos de Valle del Cauca y Cundinamarca están principalmente relacionados con el PYMV, mientras que begomovirus que afectan este mismo cultivo pero en Santander están relacionados con ToVEV, un geminivirus no emparentado con el PYMV (Guzmán et al. 1997; Argüello & Medrano, 2001; Romay et al. 2010). Asimismo de los cladogramas se puede inferir que los geminivirus que afectan al tomate en Colombia todos pertenecen al hemisferio occidental (figura 2) pues se agrupan junto con otros begomovirus reportados en esta parte del planeta tales como PHYVV, SiGMV, AbMV, PePGMV y BDMV este último ampliamente reportado afectando el cultivo de frijol en Colombia. A la fecha y de acuerdo a los resultados obtenidos en este estudio no hay evidencia de que algún geminivirus monopartita perteneciente al hemisferio oriental se encuentra afectando cultivo de tomate en Colombia (datos no mostrados).
Al realizar el análisis de secuencias de ácidos nucleicos para el extremo 5' del gen de la proteína asociada a replicación (Rep) de cada una de las clonas, se encontró la presencia de cuatro variantes virales asociadas a PYMV: una primera asociada a PYMV-Valle con valores de identidad entre 0,96 y 0,99 con los clones 6TuV-A, 7CaV-A, 28SiC-A y 30SiC-A identificados en los departamentos de Valle del Cauca y Cundinamarca; una segunda entidad viral asociada a PYMV-Ma con valores de identidad de 0,93 con los clones 58PdCS-A, 59PdC-A y 61PdCS-A identificados en el departamento Santander; una tercera entidad asociada a CdTVTo con valores de identidad de 0,82 y 0,83 con los clones 27SiC-A y 29SiC-A; y una cuarta entidad viral asociada con los begomovirus PYMV - TT y BDMV con una identidad de 0,86 con el clon 66CeS-A identificado en el departamento de Santander (Tabla 4). Es interesante encontrar tres clones, 27SiC-A, 29SiC-A y 66CeS-A, que a nivel de CP tienen una identidad de 0,94 -1,0 con PYMV, pero cuando se compara con Rep esta identidad tiene un valor bajo (Tabla 4). Este mismo resultado se encuentra cuando se compara su región intergénica (RI) encontrado un valor de identidad con PYMV de 0,47 (datos no mostrados). Con el fin de establecer la identidad de estos begomovirus, actualmente se está trabajando en el laboratorio en la obtención del genoma completo de 27SiC-A, 29SiC-A y 66CeS-A.
Análisis de interones presentes en el promotor del gen de la proteína asociada a la replicación (Rep) de los begomovirus que afectan tomate en Colombia
Los geminivirus han evolucionado gracias a eventos de recombinación y pseudorecombinación sucedidos entre ellos a lo largo de la escala evolutiva (Van der Waltz et al. 2009; Padidam et al. 1999). Ese último mecanismo evolutivo (pseudorecombinación) está determinado en gran medida por la similitudes a nivel del los elementos cis-regulatorios (particularmente los iterones) presentes en el promotor del gen de la proteína asociada a la replicación (Rep) de estos virus. Basado en la teoría del iterón (Argüello-Astorga et al. 1994; Argüello-Astoga & Ruiz-Medrano, 2001) se realizó el análisis bioinformático de los elementos cis-regulatorios (enfocado en la identificación de los iterones) presentes en el promotor del gen de la proteína asociada a la replicación (Rep) de cada uno de los aislados geminivirales que se encontraron afectando el tomate en Colombia (figura 3). La clona 59PdCS-A se descartó de este análisis dado que presenta una delección en la región común que no permite obtener resultados válidos. Por medio de este análisis se obtuvieron los siguientes resultados: primero, la disposición y número de los iterones presentes en la región promotora de Rep de los aislados geminivirales colombianos es similar a la que presentan los geminivirus presentes en el Hemisferio Occidental, de acuerdo al análisis filogenético y estructural publicado por Argüello-Astorga y colaboradores (1994). Segundo, de acuerdo a la disposición y orientación de los iterones presentes en la región promotora de Rep de los aislados geminivirales colombianos se puede hablar que éstos se distribuyen en dos grupos: un primer grupo de geminivirus (28SiC-A,30SiC-A, 6TuV-A, 7CaV-A, 58PdCS-A y 61PdCS-A) tienen cuatro iterones, dos en sentido directo y dos en sentido invertido, con una secuencia consenso AHHGGGGG; un segundo grupo de geminivirus (66CeS-A, 27SiC-A y 29SiC-A) que afectan el tomate en Colombia presentan una disposición diferente presentando únicamente tres iterones, dos en sentido directo y uno en sentido invertido, con dos secuencias consensos: ATYGGKGT para 66CeS-A y ATTSGGTR para los aislados 27SiC-A y 29SiC-A. Esta última secuencia consenso de iterones es diferente a las reportadas a la fecha en otros geminivirus del hemisferio occidental y la cuál seria un aporte novedoso de este trabajo. Tomando como base el resultado de estos análisis de iterones presentes en la región promotora de Rep de los aislados geminivirales que se encontraron afectando el tomate en Colombia se puede hipotetizar lo siguiente:
- Aquellos aislados geminivirales colombianos que tienen iterones cuyo consenso es AHHGGGGG presentes en muestras de Valle del Cauca, Santander y Cundinamarca no solo pueden sufrir eventos de pseudorecombinación entre ellos sino además con otros begomovirus cuyos iterones sean similares a nivel de secuencia tales como PYMV-Guadalupe, PYMV-Valle, ToMoTV y MalYMV. Si bien es cierto que ToMoTV, MalYMV y PYMV-Guadalupe no han sido reportados en Colombia afectando el tomate, no seria descabellado suponer que con el trasiego de material vegetal en un mundo globalizado como éste, la introducción de estos geminivirus y su posible pseudorecombinación con cualquiera de los aislados colombianos cuyos iterones sean similares podría conducir a una enfermedad geminiviral más acentuada (sinergismo) en tomate causando mayores pérdidas a los productores. Existen pruebas irrefutables que dos geminivirus pueden pseudorecombinarse gracias a que la secuencia consenso de sus iterones era similar; los estudios que llevaron a cabo Ramos y colaboradores en 2003 comprobaron, por medio de ensayos de inoculación por biobalistica, que el virus taino del tomate (ToMoTV) co-bombardeado junto con el PYMV-Guadalupe en plantas de tomate y tabaco producían la aparición de síntomas típicos de la enfermedad geminiviral. Ambos virus tenían secuencias de iterones similares y jamás se habían reportado juntos afectando el tomate. Es posible que este sinergismo entre geminivirus pueda evolutivamente permitirles la conquista de nuevos hospederos mediante una rápida adaptación a los mismos.
- En cuanto se refiere a los aislados geminivirales colombianos cuyos iterones tienen secuencias consenso ATYGGKGT y ATTSGGTR, estos no podrían pseudorecombinarse entre ellos ni con aquellos aislados geminivirales cuyo iterón tenga la secuencia consenso AHHGGGGGG. Es decir, posiblemente al encontrarse ambos aislados en una misma planta de tomate en vez de darse una relación sinérgica posiblemente se daría un fenómeno de interferencia, gracias a la diferencia entre sus iterones, debido a que competirían entre si por el replisoma de la planta. Es posible que desde un punto de vista evolutivo estos eventos de interferencia conduzcan a las poblaciones de geminivirus con iterones diferentes a evolucionar hacia nichos distintos o simplemente limiten sus posibilidades de sinergismo solamente a un grupo particular de geminivirus que están compartiendo un área biogeográfica particular. Este podría ser el caso del aislado de Santander (66CeS-A) el cuál por su disposición, secuencia y orientación de sus iterones podría pseudorecombinar con PYMV-Trinidad así como con TMoLCV-Zulia, todos los cuáles son geminivirus que están afectando el cultivo de tomate en un área geográfica común del nororiente del continente suramericano. Un caso particular se evidenció en dos aislados geminivirales que se encontraron afectando el cultivo de tomate en Cundinamarca (27SiC-A y 29SiC-A) cuya secuencia consenso de iterones era ATTSGGTR; éstos podrían formar pseudorecombinantes viables entre ellos mismos pero no con otros aislados de PYMV reportados a la fecha en el Caribe y en Colombia. Es decir, estos aislados a pesar de estar emparentados filogenéticamante con PYMV no pueden transreplicarse con otros PYMV que no tengan iterones similares en secuencia con aquellos que éstos poseen. Asimismo esta diversidad en iterones encontrados al interior del las poblaciones de PYMV que están afectando el tomate en Colombia están en perfecta concordancia con la comprobación de la presencia de infecciones mixtas de geminivirus en el cultivo de tomate en Colombia. Es decir, a través del análisis bioinformático de iterones presentes en el promotor de la proteína asociada a la replicación de geminivirus que están afectando el tomate en Colombia, fue posible encontrar evidencia adicional que corrobora los resultados que indican la presencia de infecciones mixtas de éstos afectando este cultivo en diferentes zonas geográficas del país.
Conclusiones
Los resultados obtenidos en este trabajo de investigación permiten concluir que los geminivirus que están afectando actualmente el cultivo de tomate en Colombia pertenecen al género de los Begomovirus y todos son bipartitas. No hay evidencia a la fecha de begomovirus monopartitas afectando esta hortaliza en Colombia. Se encontró que variantes de los begomovirus identificados afectando el tomate en campo están relacionados filogenéticamente con el Virus del mosaico amarillo de la papa (PYMV) y con el Tomato venezuela virus (ToVEV). Existe una distribución geográfica de variantes de begomovirus identificados afectando el tomate en campo en Colombia: en el departamento de Santander predominan geminivirus relacionados filogenéticamente con PYMV-Martinica y con ToVEV; en el departamento de Cundinamarca hay begomovirus emparentados con PYMV-Valle y con el ToVEV; y en el departamento de Valle del Cauca predominan únicamente geminivirus relacionados con el PYMV-Valle.
Hay evidencia que indica que la enfermedad geminiviral en tomate en Colombia es causada por el accionar de dos o más geminivirus en infecciones mixtas y que este hecho puede explicar la presencia de síntomas severos de enfermedad viral en algunos de las zonas tomateras muestreadas en el País. La evidencia de la presencia de infecciones mixtas proviene de los resultados obtenidos por medio de RFLP, secuenciación de fragmentos de PCR así como análisis de Iterones presentes en el promotor del la proteína asociada a la replicación de geminivirus (Rep/ AL1). El análisis bioinformático de Iterones presentes en el promotor de la proteína asociada a la replicación de geminivirus (Rep) permite hipotetizar los siguiente: algunas de las variantes geminivirales de Valle del Cauca, Santander y Cundinamarca pueden sufrir eventos de pseudorecombinación entre ellos así como con los begomovirus PYMV-Guadalupe, PYMV-Valle, ToMoTV y MalYMV; mientras que dos variantes geminivirales encontradas en Cundinamarca solo podrían formar pseudorecombinantes viables entre ellos mismos pero no con otros aislados de PYMV reportados a la fecha en el Caribe y en Colombia. Estas interacciones pueden conducir a sinergismos o a interferencias según la identidad a nivel de secuencia que se pueda verificar por sus iterones.
El que los geminivirus que se encontraron afectando el tomate en Colombia sean en su mayoría variantes begomovirales de PYMV facilita implementar un programa de mejoramiento genético por vías tradicionales o biotecnológicas encaminado a la búsqueda de materiales de tomate con resistencia derivada de este fitopatógeno. A este respecto, nuestro grupo de trabajo se encuentra actualmente evaluando un banco de germoplasma de tomate silvestre inoculando aislados geminivirales colombianos mediante biobalística para identificar materiales promisorios resistentes a estos virus endémicos de Colombia.
Agradecimientos
El presente proyecto de investigación fue financiado con recursos del Ministerio de Agricultura y Desarrollo Rural de Colombia, contrato 134-2008N6396-3460, y se desarrollo en asocio con la empresa Defrescura S.A. y la Universidad Nacional de Colombia. Los autores expresan su mas sincero agradecimiento a la Compañía Agroindustrial de Semillas S.A., por su invaluable ayuda para la organización y recolección del material vegetal a lo largo ancho del territorio Colombiano. Betancurt-Perez J. agradece a Colciencias la beca recibida para realizar estudios de doctorado en Ciencias Agropecuarias.
Referencias bibliográficas
1 Argüello-Astorga, G. R., and R. Ruiz-Medrano. 2001. An iteron-related domain is associated to motif 1 in the replication proteins of geminiviruses: identification of potential interacting amino acid-base pairs by a comparative approach. Arch. Virol. 146:1465-1485.
2 Argüello-Astorga, G., Guevara-Gonzalez, R. G., Herrera-Estrella, L. R., and R. F. Rivera-Bustamante. 1994.Geminivirus replication origins have a group-specific organization of iterative elements: a model for replication.Virol. 203: 90-100.
3 Benson D. A., Karsch-Mizrachi I., Lipman D.J., Ostell J. and E. W. Sayers. 2011. Genbank. Nucleic Acids Res. 39: on line.
4 Brown, J. K., Idris, A. M., Torres-Jerez, I., Banks, G. K., and S. D. Wyatt. 2001. The core region of the coat protein gene is highly useful in establishing the provisional identification and classification of begomoviruses. Arch. Virol. 146: 1581-1598.
5 Brown, J. K., Idris, A. M., Ostrow, N., Goldberg, R. F. and D.C. Stenger. 2005. Genetic and phenotypic variation of Pepper golden mosaic virus complex. Phytopathol. 95: 1217-1224.
6 Coutts, R. H., Coffin, R. S., Roberts, E. J. and W. D. Hamilton. 1991. The nucleotide sequence of the infectious cloned DNA components of potato yellow mosaic virus. J. Gen. Virol. 72: 1515-1520.
7 Duffy, S., and E. C. Holmes. 2009. Validation of high rates of nucleotide substitution in geminiviruses: phylogenetic evidence from East African cassava mosaic viruses. J. Gen. Virol. 90:1539-1547.
8 FAOSTAT. 2007. Food and Agriculture Organization of the United Nations. http://faostat.fao.org/.
9 Fauquet, C. M., Bisaro, D. M., Briddon, R. W., Brown, J. K., Harrison, B. D., Rybicki, E. P., Stenger, D. C., and J. Stanley. 2003. Revision of taxonomic criteria for species demarcation in the Geminiviridae, and an updated list of begomovirus species. Arch. Virol. 148:405-421.
10 Fauquet, C. M., Briddon, R. W., Brown, J. K., Moriones, E., Stanley, J., Zerbini, M. and X. Zhou. 2008.Geminivirus strain demarcation and nomenclature. Arch Virol. 153:783-821.
11 Ge, L., Zhang, J., Zhou, X., and Li, Hongye. 2007. Genetic Structure and Population Variability of Tomato Yellow Leaf Curl China Virus. J. Gen. Virol. 34: 5902-5907.
12 Gutiérrez, C. 2000. DNA replication and cell cycle in plants: learning from geminiviruses. The EMBO J. 19: 792-799.
13 Guzman, P., Arredondo, C. R., Emmatty, D., Portillo, R. J. and R. L. Gilberton. 1997. Partial characterization of two whitefly-transmitted geminiviruses infecting tomatoes in Venezuela. Plant Dis. 81: 312.
14 Hall, T.A. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl. Acids. Symp. Ser. 41:95-98.
15 Hanley-Bowdoin, L., Settlage, S. B., Orozco, B. M., Nagar, S. and D. Robertson. 1999. Geminiviruses: Models for plant DNA replication, transcription, and cell cycle regulation. Crit. Rev. Plant. Sci. 18:71-106.
16 Harrison, B.D., Zhou, X., Otim-Nape, G.W., Liu, Y. and D. Robinson. 1997. Role of a novel type of double infection in the geminivirus induced epidemic of severe cassava mosaic in Uganda. Ann. Appl.Biol. 131: 437-448.
17 Mahy, B. W. and M. H. V. Van Regenmortel. 2010. Plant and fungal virology. Elsevier. p 109.
18 Martínez, A. K., Morales, F. J. y F. A. Vallejo. 2008. Caracterización molecular de un begomovirus del tomate en el Valle del Cauca, Colombia, y búsqueda de fuentes de resistencia para el mejoramiento de la variedad Unapal Maravilla. Acta Agron. 57: 167-173.
19 Mendez-Lozano, J., Torres-Pacheco, I, Fauquet, C.M., and R.F. Rivera-Bustamante. 2003. Interactions between geminiviruses in a naturally occurring mixture: Pepper huasteco virus and Pepper golden mosaic virus.Phytopathol. 93: 270-277.
20 Morales, F. J., and P. K. Anderson. 2001. The emergence and dissemination of whitefly-transmitted geminivirus in Latin America. Arch.Virol. 146:415-441.
21 Morales, F. J., Martínez, A.K. y A. C. Velasco. 2002. Nuevos brotes de geminivirus en Colombia. Fitopatol. Colombiana. 26:76-78.
22 Morales, F. y C. Castaño. 2008. Enfermedades virales del frijol común en America Latina. CIAT-publicaciones. Palmira (Valle-Col). 86p.
23 Mubin, M., Tahir, M. N., Shahid, M. S., Briddon, R. W., and S. Mansoor. 2009. Characterization of begomovirus components from a weed suggests that begomoviruses may associate with distinct DNA satellites in different geographical locations. Virus Genes 40:452-457.
24 NCBI (http://www.ncbi.nlm.nih.gov).
25 Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals.Genetics 89: 583-590.
26 Padidam, M., Beachy, R. N., and C. M. Fauquet. 1995. Classification and identification of geminiviruses using sequence comparisons. J. Gen. Virol. 76: 249-263.
27 Padidam. M., Sawyer. S. and C. M. Fauquet. 1999. Possible emergence of new geminiviruses by frequent recombination. Virol. 265:218-224.
28 Pita, J. S., Fondong, V. N., Sangare, A., Otim-Nape, G. W., Orwal, S., and C.M. Fauquet. 2001. Recombination, pseudorecombination, and synergism are determinant keys to the epidemic of severe cassava mosaic diseases in Uganda. J. Gen. Virol. 82: 655-665.
29 Polston, J. E., and P. K. Anderson. 1997. The emergence of whitefly-transmited geminivirus in tomato in the western hemisphere. Plant Dis. 81:1358-1369.
30 Ribeiro, S. G., Ambrozevicius, L. P., Avila, A. C., Bezerra, I. C., Calegario, R. F., Fernandez, J. J., Lima, M. F., Mello, R. N., Rocha, H. and F. M. Zerbini. 2003. Distribution and genetic diversity of tomato-infecting begomoviruses in Brazil. Arch.Virol. 148: 281-295.
31 Rojas M. R., Gilbertson R. L., Ruseel D. R. and D.P. Maxwell. 1993. Use of generate oligonucleótidos in the polymerase chain reaction to detect whitefly-transmitted geminivirus. Plant dis. 77:340-347.
32 Rojas, M. R.; Hagen, C.; Lucas, W.J. and R. L. Gilbertson. 2005. Exploiting chinks in the plants armor: Evolution and emergence of Geminivirus. Annu. Rev. Phytopathol. 43: 361-394.
33 Romay, G., Geraud-Pouey, F., Chirinos, D., Morales, F., Herrera, E., Fernandez, C. and A. Martinez. 2010. Transmission of Tomato Venezuela Virus by Bemisia tabaci (Gennadius) (Hemiptera: Aleyrodidae), in Maracaibo, Venezuela. Neotrop Entomol. 39:266-274.
34 Sambrook, J and D. Russell. 2001. Molecular Cloning. A laboratory Manual. 3rd. New York: Cold Spring Harbor Laboratory. 2344 p.
35 Sanz, A. I., Fraile, A., Garcia-Arenal, F., Zhou, X., Robinson, D.J., Khalid, S., Butt, T. and B.D. Harrison. 2000. Multiple infection, recombination, and genome relationship among begomovirus isolates found in cotton and others plants in Pakistan. J. Gen.Virol. 81: 1839-1849.
36 Seal, S. E., Van den Bosch, F. and M. Jeger. J. 2006. Factors influencing begomovirus evolution and their Increasing global significance: implications for sustainable control. Crit. Rev. Plant. Sci. 25: 1-23.
37 Tamayo P. J. y J. E. Jaramillo. 2006. Enfermedades del tomate, pimentón, ají y berenjena en Colombia. Guía para su diagnóstico y Manejo. Corpoica. pp. 69-79.
38 Tamura, K., Peterson, D., Peterson, N., Stecher, G., Nei, M., and S. Kumar. 2011. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods.Mol. Biol. Evol. 28: 2731-2739.
39 Thompson, J. D., Gibson, T. J., Plewniak, F., Jeanmougin, F. and D. G. Higgins. 1997. The CUSTALX windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acid Res. 25:4876-4882.
40 Ueda, S., Onuki, M., Hanada, K. and Y. Takanami. 2008. Unique grouping of the Far East Asian begomovirus complex based on sequence analyses of the DNA-A genome and associated DNAb satellite molecules isolated from tomato, honeysuckle and Eupatorium plants in Japan. Arch Virol 153:417-426.
41 Urbino, C., Polston, J. E., Patte, C. P., and M. L. Caruana. 2004. Characterization and genetic diversity of Potato yellow mosaic virus from the Caribbean. Arch Virol. 149: 417-424.
42 Uribe-Echeverri, P. T; Lopez-Lopez, K and J. C. Vaca-Vaca. 2011. Identificación molecular de virus RNA que están afectando el cultivo de tomate en el departamento del Valle del Cauca- Colombia. XXX Congreso Colombiano y XVI Latinoamericano de Fitopatología. Bogota D.C. Colombia. 17, 18 y 19 de Agosto del 2011.
43 Vaca-Vaca, J. V., Betancurt-Pérez, J. F., y López-López. K. 2011. Detección, identificación y localización geográfica de Begomovirus que afectan al tomate en Colombia. Rev. Colomb. Biotecnol. 8: 115-122.
44 Willment, J. A., Martin, D. P., and E. P. Rybicki. 2001. Analysis of the diversity of African streak mastreviruses using PCR-generated RFLPs and partial sequence data. Journal of Virol. Methods 93: 75-87.
44 Wyatt, S.D. and J.K. Brown. 1997. Detection of geminiviruses in aqueous extracts by polymerase. Phytopathol. 86: 1288-1293.
45 Zhang, Z., Schwartz, S., Wagner, L., and W. Miller. 2000. A greedy algorithm for aligning DNA sequences. J Comput Biol. 7:203-14.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2012 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).