Evaluación de la presencia del gen mer A implicado en la detoxificación de mercurio a partir de actinomicetos nativos del humedal de La Conejera
Palabras clave:
Streptomyces, mercurio-resistencia, gen mer A, plásmidos, humedales, mercury-resistance, mer A gene, plasmid, wetlands (es)Evaluación de la presencia del gen mer A implicado en la detoxificación de mercurio a partir de actinomicetos nativos del humedal de La Conejera
Evaluating the presence of the mer A gene implied in mercury detoxification from native actinomycete strains from La Conejera wetlands
Carolina Rueda 1 , Marcia Aikawa 2 , Luis Daniel Prada3 , Marcela Franco-Correa4 , María Martínez4 , Gabriel Padilla5
1Microbióloga Industrial, Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá. crueda@javeriana.edu.co2Universidad de São Paulo, Instituto de Pesquisas Biomedicas, Brasil.maikawa@icb.usp.br
3Joven Investigador, Departamento de Microbiología, Facultad de Ciencias, Grupo Unidia, Pontificia Universidad Javeriana, Bogotá. lprada@javeriana.edu.co
4Profesor, Departamento de Microbiología, Facultad de Ciencias, Grupo de Biotecnología Ambiental e Industrial, Pontificia Universidad Javeriana, Bogotá. franco@javeriana.edu.co y mmmartin@javeriana.edu.co
5Profesor-investigador, Universidad de São Paulo, Instituto de Pesquisas Biomedicas, Brasil. gpadilla@icb.usp.br
Recibido: agosto 21 de 2009 Aprobado: noviembre 23 de 2009
Resumen
Se aislaron diez cepas de actinomicetos nativos del humedal La Conejera a partir de muestras de sedimento y agua, con el fin de determinar la capacidad de resistencia y detoxificación de mercurio, evaluando la presencia del gen mer A involucrado en el proceso. La prueba de resistencia fue realizada mediante un test de sensibilidad tomando concentraciones desde 3,68x10-3 mM hasta 10 mM de HgCl2. Se realizaron curvas de crecimiento de 192 h para las cepas resistentes al mercurio, en un medio suplementado con 0,01 mM y 0,05 mM de HgCl2, realizando una fermentación discontinua y midiendo el crecimiento por la técnica de peso seco. A partir de estos resultados se determinó un tiempo de adaptación de 24 h y una producción máxima de biomasa de 2,72 g·L-1 a la hora 72. Paralelamente, se realizó la secuenciación de los genes de resistencia al mercurio de Streptomyces lividans 1326, por medio del diseño de los oligonucleótidos capaces de amplificar el extremos 3 del gen mer A, codificador de la enzima mercurio reductasa. Se optimizaron las condiciones de amplificación por PCR para obtener un producto amplificado de 686 pb aproximadamente. Finalmente, se realizó una electroforesis en campo pulsado comprobando la presencia de plásmidos en la cepa KH7 con tamaños aproximados de 50, 90 y 300 Kb, no integrados al cromosoma y posiblemente asociados a la resistencia presentada frente al metal.
Palabras clave: Streptomyces, mercurio-resistencia, gen mer A , plásmidos, humedales.
Abstract
Ten native actinomycete strains were isolated from water and sediment samples collected in La Conejera wetlands to assess mercury resistance and detoxification ability for evaluating the presence of the mer A gene involved in that process. Sensitivity assays were performed using 3.68 x 10-3 mM to 10 mM HgCl2 concentrations. Growth curves were generated for each resistant strain after 192 h in discontinuous fermentation in medium supplemented with 0.01 mM and 0.05 mM HgCl2. Biomass growth was then measured by dry weight, maximum value (2.37 gL-1) being obtained after 72 h. Primer pairs were designed based on the sequence encoding the mercury-reductase enzyme in Streptomyces lividans 1326. PCR conditions for amplifying that region were standardised. The mer A gene was identified in the KH7 strain, resulting in an amplified product of around 686 bp. The KH7 strain electrophoresis profile obtained by pulsed field gel electrophoresis showed 50, 90 and 300 Kb plasmid DNA which could be related to metal resistance in this strain.
Key words: Streptomyces, mercury-resistance, mer A gene, plasmid, wetlands.
Introducción
El mercurio es uno de los metales que por su toxicidad representa gran peligro para el medioambiente. Su entrada al medio es causada por procesos naturales o antropogénicos; este último debido al uso indiscriminado de compuestos mercuriales en la industria y la agricultura, así como a la falta de control de afluentes contaminados como sucede en los humedales, lo cual ocasiona daños irreversibles tanto para la biota terrestre como acuática. Los actinomicetos son bacterias Gram positivas que presentan crecimiento filamentoso y alto contenido GC en su ADN; la resistencia de estas bacterias frente a los metales no ha sido investigada con profundidad a pesar de ser metabólica y biosintéticamente versátiles.
Nakahara y et al. (1985) fueron los primeros en estudiar la resistencia frente al mercurio en actinomicetos, la cual fue inducida en Streptomyces lividans 1326, con la posterior clonación y análisis del operón responsable de ésta (Eichenseer y Altenbuchener, 1994). Los genes de resistencia al mercurio en Streptomyces lividans 1326 fueron localizados en un elemento amplificable AUD2, con características semejantes a las de un transposón con secuencias de inserción repetidas al final, debido seguramente a la inestabilidad genética (Eichenseer y Altenbuchner, 1994; Osborn et al., 1997). Streptomyces lividans presenta su resistencia frente al mercurio mediante seis fragmentos abiertos de lectura (ORF) organizados en dos operones de transcripción divergentes. Los genes de regulación y de trasporte forman un operón y las enzimas mercurio reductasa y organomercurio liasa forman el segundo (Brünker et al., 1996).
El gen mer B codifica para la organomercurio liasa la cual rompe los enlaces C-Hg, liberando iones de Hg+2, los cuales se reducen a una forma volátil, el mercurio elemental (Hg0), por la acción de la mercurio reductasa (mer A) (Sedlmer y Altenbuchener, 1992; Rother et al., 1999). Por lo anterior, a partir de los aislamientos de actinomicetos nativos del humedal La Conejera en Bogotá, Colombia, se pretendió evaluar la presencia del genmer A involucrado en la reducción de Hg+2 hasta Hg0.
Materiales y métodos
Muestreo
Diez muestras de sedimento (lodos) y agua, procedentes del humedal La Conejera, en el punto de descarga de los efluentes provenientes de cultivos de flores y de residuos hospitalarios, fueron colectadas a 30 cm de profundidad en recipientes estériles, y transportadas hasta el laboratorio en condiciones de refrigeración. Una vez en el laboratorio fueron procesadas, realizando diluciones seriadas en agua peptonada estéril (0,1% p/v) tomando de cada dilución 0,1 ml para la siembra en superficie sobre placas de agar avena (Thumar y Singh, 2007; Jayasinghe y Parkinson, 2008). Igualmente, fueron recuperadas muestras liofilizadas de actinomicetos nativos aislados del humedal, conservadas en el laboratorio de Microbiología Ambiental y de Suelos de la Pontificia Universidad Javeriana, codificadas como KH1, KH3, KH7, KH16 y AG4, AG5 y AG14.
Ensayos de sensibilidad
El ensayo de sensibilidad fue realizado utilizando el método de anillos de vidrio descrito por Gauze (1965) en placas de agar avena y medio ISP2 (g·L-1: glucosa 4, extracto de malta 10, extracto de levadura 4, agar: 15) (Difco Laboratorios, Detroit, MI, USA) las cuales fueron sembradas e incubadas a 28 °C durante 5 días, teniendo en cuenta como parámetro de selección la formación de halo de menor tamaño alrededor de los anillos. Para este ensayo se utilizaron diferentes concentraciones del metal (10; 5; 3; 2,5; 2; 1,5; 1; 0,5; 0,1 y 0,05 mM) con el fin de determinar la concentración mínima inhibitoria y la máxima tolerable, y de esta forma definir las concentraciones con las cuales se realizarían los siguientes ensayos de la cinética. Se realizaron 5 réplicas por tratamiento.
El crecimiento de las cepas fue comparado usando como control positivo S. lividans 1326 y S. avermitilis cepa sensible al metal (Nakahara et al., 1985), obtenidos del laboratorio de Genética Microbiana del Instituto de Ciencias Biomédicas, Universidad de São Paulo, Brasil.
Cinética de crecimiento
La velocidad de crecimiento de las cepas seleccionadas se determinó mediante la técnica de peso seco a partir de una fermentación discontinua en erlenmeyer de 1000 ml, con 200 ml de medio líquido ISP2 suplementado con mercurio (0,01 y 0,05 mM de HgCl2) en agitación constante (250 rpm) a 28 °C, tomando alícuotas de 10 ml cada 24 h durante 8 días. De esta forma, se determinó el tiempo de adaptación de los microorganismos frente al metal y la cepa con mayor producción de biomasa. Para este ensayo se realizaron 5 réplicas.
Diseño de los oligonucleótidos
Con base en la secuenciación de los genes de resistencia al mercurio de S. lividans 1326 (gen mer A) (Sedlmeier y Altenbuchner, 1992), y con la ayuda del programa Oligos (National Biosciences, Inc, Pymouth MN) se diseñaron unos oligonucleótidos capaces de amplificar la secuencia del gen mer A en las cepas mercurio resistentes. La síntesis de los mismos fue realizada por Invitrogen Life Technologies (São Paulo, Brasil).
Extracción de ADN
Se inocularon las cepas en medio YEME (g·L-1:glucosa 5; extracto de levadura 1,5; bacto pectona 2,5; extracto de malta 1,5; sacarosa 17; MgCl2 (2,5 M) 500 µl; Glicina 20% p/v) que fueron incubadas durante 3 días a 28 °C y 200 rpm. Obtenida la biomasa, se centrifugó el medio para obtener las células, las cuales fueron lavadas con Suc-TE, posteriormente se incubaron con lisozima (5 mg·mL-1) durante 1 h a 37 °C, transcurrido el tiempo se adicionó SDS al 1% p/v (0,7 mL) y EDTA 0,5M (1,2 mL). El producto lisado fue digerido con proteinasa K (1mg·mL-1) y tratado con fenol, fenol-cloroformo (25:24) y solución de cloroformo-alcoholisoamílico (24:1). El ADN fue precipitado con alcohol isopropílico mezclando por inversión (Kieser et al., 2000).
Optimización de las condiciones de la PCR
Las condiciones iniciales para la PCR se basaron en la metodología utilizada por Metsa- Ketela et al. (1999), tomando 10 ng de ADN genómico de S. lividans 1326, 100 pMol de cada primer, 1 mM dNTPS, 2 U Vent Polymerasa (New England Bioabs), 1x Thermopol Buffer (10 mM KCl; 20 mM Tris-HCl pH 8,8; 10 mM (NH4)SO4; 2 mM MgSO4; 0,1% p/v Triton X-100). Manteniendo un volumen total de 50 µl, con ciclos de: 1 ciclo: 96 °C durante 2 min, 56 °C durante 2 min, 73 °C durante 1,5 min; 28 ciclos: 96 °C, 1 min; 56 °C, 2 min; 73 °C, 1,5 min y 1 ciclo: 96 °C, 1 min; 56 °C, 2 min; 73 °C, 8,5 min.
El producto de la PCR fue visualizado en una electroforesis utilizando como tampón de corrida TAE 0,1% p/v (tris base, ácido acético, EDTA pH 8,0) y gel de agarosa al 0,8% p/v. La concentración de ADN para cada muestra fue de 10 µg L-1 respectivamente. El ADN fue separado a 60V durante 180 min.
PFGE (Pulsed-Field gel electrophoresis)
Las cepas resistentes a mercurio fueron incubadas en medio YEME durante 4 días y centrifugadas a 8000 rpm a 5 °C durante 15 min. El precipitado fue suspendido en 5 ml de SucTe (g·L-1: 1,51 Tris-HCl pH 8,0; 4,65 EDTA pH 8,0; 51,35 sacarosa) y diluido hasta obtener una densidad óptica (DO600) de 1,8-2,2 nm de la suspensión celular, con base en la metodología de Kieser et al. (2000) recomendada para bacterias filamentosas. Un volumen de 400 µl de la suspensión bacteriana fue mezclado con 800 µl de la solución 2,0% p/v de agarosa mantenida a 50 °C. Una vez solidificada la solución, los bloques formados fueron transferidos a tubos Falcon con 10 ml de SucTe, y se adicionó lisozima (2mg·mL-1) durante 12 h a 37 °C. Transcurrido el tiempo, los bloques se trataron con DNS durante 24 h a 50 °C. Después de este periodo fueron lavados con tampón TE y tratados con PMFS (40µL·mL-1) dos veces a 4 °C. Finalmente, fueron lavados con tampón TE y mantenidos a 4 °C (Kieser et al., 2000). Las condiciones de corrida fueron de 6s (6,0 v·cm-1 a 14°C) durante 24 h, 70 s (200 v·cm-1 a 14°C) por 15 h y 120 s (200 v·cm-1 a 14°C) durante 11 h con buffer TBE 0,5% v/v (Ravel et al., 1998).
Análisis estadístico
Los resultados obtenidos en las pruebas descritas como parámetro para la selección de las cepas mercurio resistentes fueron analizados con el 95% de confianza, mediante un análisis de varianza bifactorial para posteriormente determinar la homogeneidad entre los grupos según Scheffe, postulando hipótesis nulas donde las diferentes concentraciones de mercurio y las cepas evaluadas producen un mismo halo de sensibilidad (Wayne, 2008).
Resultados y discusión
Diez cepas de actinomicetos fueron aisladas en dos zonas del humedal La Conejera, obteniéndose el mayor número de aislamientos en la zona correspondiente a efluentes procedentes de cultivos de flores. De igual manera, un total de 7 cepas nativas del humedal fueron recuperadas a partir de liofilizados del laboratorio de Microbiología Ambiental y de Suelos de la Pontificia Universidad Javeriana.
Una vez realizado el test de sensibilidad los resultados de las repeticiones se promediaron, dando un solo resultado representativo de las cepas para cada microorganismo enfrentado a las diferentes concentraciones del metal (figura 1). A partir de este resultado las cepas con menor halo de sensibilidad fueron identificadas como KH7 y CR11 respectivamente.
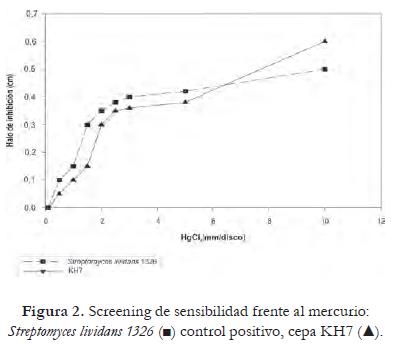
Las cepas evaluadas y las concentraciones propuestas de HgCl2 presentaron diferencias significativas (P=0,05) en la formación de halos de resistencia, parámetro que determinó el rechazo de las hipótesis nulas propuestas en el estudio. La cepa KH7 demostró la mayor resistencia frente al HgCl2 con resultados similares de resistencia comparada con el control positivo S. lividans 1326 (figura 2) tolerando todas las concentraciones propuestas desde 0,05 a 10 mM con halos inferiores a 6 mm. Los resultados anteriores proponen la relación entre la resistencia al HgCl2 y la actividad de la enzima mercurio reductasa codificada por los genes mer A. La cepa KH7 muestra una fase logarítmica hasta la hora 72 seguida de la fase estacionaria hasta la hora 120; la curva control presenta un comportamiento similar con una fase logarítmica hasta la hora 120 y producción máxima de biomasa de 2,93 g·L-1. Hay similitudes con la curva control, sin afectar la actividad enzimática y el crecimiento del microorganismo (figura 3). A una concentración de 0,05 mM de HgCl2 se presenta una marcada disminución de la biomasa celular (0,73 g·L-1) determinada por el efecto tóxico del HgCl-, al compararla con la concentración de 0,01 mM donde se obtuvo como valor máximo de producción de biomasa 3,10 g·L-1 en la hora 96. Esto se puede explicar por la formación de enlaces covalentes con los grupos sulfhidrilo de la cisteína presentes en las proteínas de la pared y membrana celular, siendo posiblemente la causa de la baja cinética registrada.
En relación con la secuenciación del gen de resistencia al mercurio de Streptomyces lividans 1326 (gen mer A), a partir del cual se diseñaron los oligonucleótidos capaces de amplificar los extremos 3 del gen mer A, permitió que se cumplieran las variables propuestas para el éxito en la amplificación. Los oligonucleótidos construidos correspondieron al oligonucleótido forward secuencia (5 a 3): GAC CTC CAC CAC CGC CAT G. La secuencia del oligonucleótido reverso fue (5 a 3): GGG ACA GGG TGC GGG TCT G. Las condiciones de corrida para la PCR fueron optimizadas y estandarizadas para la amplificación del gen mer A encargado de la síntesis de la enzima mercurio reductasa.
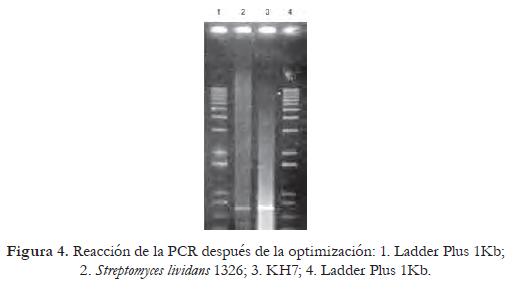
Con una disminución de la temperatura de anillaje de 56 °C a 50 °C y utilizando una temperatura de extensión para cada ciclo de 68 °C, se logró obtener un fragmento amplificado de aproximadamente 686 pb (figura 4) correspondiente al gen mer A manteniéndose el resto del programa igual al establecido previamente.
Con la realización de la PFGE fue determinada la presencia de tres plásmidos naturales lineales, detectados en la cepa KH7 con un tamaño aproximado de 50, 90 y 300 Kb no integrados al cromosoma (figura 5). La migración de los plásmidos fue determinada de acuerdo con los diferentes pulsos de corrida establecidos en la metodología, lo cual indicó que los tres plásmidos son lineales y no circulares al poseer proteínas ligadas covalentemente a las extremidades 5 del ADN. La migración de los plásmidos fue posible solo al tratar el ADN con proteinasa K, usada después de causar la lisis celular, para ayudar a liberar el ADN de los restos de célula y probablemente eliminar los enlaces proteicos ligados a los plásmidos (Ravel et al., 1998). Con relación al ADN de S. lividans 1326, fue imposible obtener fragmentos claros asociados a los plásmidos, posiblemente derivados de la susceptibilidad que se puede presentar frente a la degradación del ADN durante la corrida de la PFGE.
En los actinomicetos, la síntesis de la enzima mercurio reductasa se asocia con la presencia de un plásmido lineal gigante pbd2 en Rhodococcus erythropolis (Dabrock, 1994; Huddleston et al., 2006) y los prj3h, pRJ3L y prj28 encontrados en especies relacionadas con S. pseudogriseolus y S. tendae (Ravel et al., 1998) donde se ha demostrado la presencia de genes mer involucrados en la remoción del mercurio que codifican para enzimas intracelulares como la Hg reductasa y la organomercurioliasa. El Hg+2 es ligado a grupos sulfhidrilo de la cisteína adyacente al sitio activo de la enzima reductasa, reduciendo dicho ión a Hg0, siendo menos tóxico y difusible al exterior de la célula (Williams, 1997).
Aunque es común encontrar estos plásmidos lineales gigantes en las especies de Streptomyces, muchas de sus funciones son desconocidas hasta ahora. Diversos plásmidos lineares se han asociado a fenotipos específicos, incluyendo sistemas de restricción y modificación (Zotchev y Schrempf, 1993), producción de antibióticos, resistencia a metales pesados y habilidad en la trasformación de productos xenobióticos (Meinhardt et al., 1997; Mochizuki et al., 2003).
Conclusión
En conclusión, la actividad metabólica de la cepa KH7, permite la reducción del Hg+2 a mercurio Hgo, al presentar en su genoma genes encargados de la función codificadora para la enzima mercurio reductasa. Lo anterior se vio reflejado en que bajas concentraciones de HgCl2 (0,01 mM) no alteraron el adecuado desarrollo reflejado en la cinética de crecimiento de los actinomicetos mercurio-resistentes, y en que se consiguió la amplificación de un fragmento de 686 pb correspondiente al extremo 3´ del merA involucrado en la reducción del mercurio. De esta manera, se permite inferir que la diversidad metabólica de estos microorganismos hace de ellos candidatos importantes para su uso en biorremediación.
Agradecimientos
A la Pontificia Universidad Javeriana por la financiación del proyecto, a la Universidad de São Paulo, Instituto de Ciencia Biomédicas en Brasil por la aceptación de la pasantía y asesoría en Biología Molecular.
Referencias bibliográficas
1 Amoroso, M. J., Castro, G. R., Carlino, F. J., Romero, N. C., Hill, R. T., Oliver, G. 1998. Screening of heavy metaltolerant actinomycetes isolated from the Salí River. Journal of General and Applied Microbiology 44: 129- 132.
2 Brünker, P., Rother, D., Sedlmeier, R., Klein, J., Mattes, R., Altenbuchner, J., Sedlmeier, R. 1996. Regulation of the operon responsible for broad-spectrum mercury resistance in Streptomyces lividans 1326. Molecular and General Genetics 251: 307-15.
3 Dadrock, B. 1994. Identification and characterization of a transmissible linear plasmid from Rhodococcus erythopolis BD2 that encodes isopropylbenzene and trichloroethane catabolism. Applied and Environmental Microbiology 60: 853-860.
4 Einchenseer, C., Altenbuchner, B. 1994. The very large amplifiable element AUD2 from Streptomyces lividans 66 has insertion sequence-like repeat at its ends. Journal of Bacteriology 176: 7107-12.
5 Gauze, G. F. 1965. Problems in the classification of antagonist of Actinomycetes. La Habana, Cuba: Ed. Medriz- Mosco, p. 22.
6 Huddleston, J. R., Zak, J. C., Jeter, R. M. 2006. Antimicrobial Susceptibilities of Aeromonas spp. Isolated from Environmental Sources. Applied and Environmental Microbiology 72: 7036-7042.
7 Jayasinghe, D. B. A. T., Parkinson, D. 2008. Actinomycetes as antagonists of litter decomposer fungi. Applied and Soil Ecology 38: 109-118.
8 Kieser, T., Bibb, M. J., Buttner, M. J., Chater, K. F., Hopwood, D. A. 2000. Practical Streptomyces genetics. United Kingdom: John Innes Foundation, Norwich. pp. 161-211.
9 Meinhardt, F., Schaffrath, R., Larsen, M. 1997. Microbial linear plasmids. Applied Microbiology and Biotechnology 47: 329-336.
10 Metsa-Ketela, M., Salo, V., Halo, L., Hautala, J., Hakala, J., Mantsala, P., Ylihonko, K. 1999. An efficient approach for screening minimal PKS genes from Streptomyces. FEMS Microbiology Letters 180: 1-6.
11 Mochizuki, S., Hiratsu, K., Suwa, M., Ishii, T., Sugino, F., Yamada, K., Kinashi, H. 2003. The large linear plasmid pSLA2-L of Streptomyces rochei has an unusually condensed gene organization for secondary metabolism. Molecular Microbiology 48: 1501-1510.
12 Nakahara, H., Schottel, J., Yamada, T., Mikakawa, Y., Asakawa, M., Harville, J., Silver, S. 1985. Mercuric reductase enzymes from Streptomyces species and group B Streptococcus. Journal of General Microbiology 132: 1053-1059.
13 Osborn, A. M., Bruce, K. D., Strike, P., Ritchie, D. A. 1997. Distribution, diversity and evolution of the bacterial mercury resistance (mer) operon. FEMS Microbiology Reviews 19: 239-262.
14 Ravel, J., Schrempf, H., Hill, R. 1998. Mercury resistance is encoded by transferable giant linear plasmids in two Chesapeake Bay Streptomyces strains. Applied and Environmental Microbiology 64 (9): 3383-3388.
15 Rother, D., Mattes, R., Altenbuchner, J. 1999. Purification and characterization of MerR, the regulator of the broad-spectrum mercury resistance genes in Streptomyces lividans 1326. Molecular and General Genetics 262: 154-162.
16 Sedlmeier, R., Altenbuchner, J. 1992. Cloning and DNA sequence analysis of the mercury resistance genes ofStreptomyces lividans. Molecular and General Genetics 236: 76-85.
17 Silver, S., Misra, T. K. 1998. Plasmid-Mediated Heavy Metal resistences. Applied and Environmental Microbiology 42: 717-743.
18 Thumar, J., Singh, S. P. 2007. Two-step purification of a highly thermostable alkaline protease from salt-tolerant alkaliphilic Streptomyces clavuligerus strain Mit-1. Journal of Chromatography B 854:198-203.
19 Wayne, D. 2008. Bioestadística: base para el análisis de las ciencias de la salud. 5 ed. México: Limusa. pp. 345- 446.
20 Williams, J. 1997. Bacterial resistance and detoxification of heavy metals. Enzyme and Microbial Technology 18 (6): 85-90.
21 Zotchev, S., Schrempf, C. 1993. The linear Streptomyces plasmid pbl1: analyses of transfer functions. Molecular and General Genetics 242: 374-38.
Evaluación de la presencia del gen mer A implicado en la detoxificación de mercurio a partir de actinomicetos nativos del humedal de La Conejera
Evaluating the presence of the mer A gene implied in mercury detoxification from native actinomycete strains from La Conejera wetlands
Carolina Rueda 1 , Marcia Aikawa 2 , Luis Daniel Prada3 , Marcela Franco-Correa4 , María Martínez4 , Gabriel Padilla5
1Microbióloga Industrial, Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá. crueda@javeriana.edu.co2Universidad de São Paulo, Instituto de Pesquisas Biomedicas, Brasil.maikawa@icb.usp.br
3Joven Investigador, Departamento de Microbiología, Facultad de Ciencias, Grupo Unidia, Pontificia Universidad Javeriana, Bogotá. lprada@javeriana.edu.co
4Profesor, Departamento de Microbiología, Facultad de Ciencias, Grupo de Biotecnología Ambiental e Industrial, Pontificia Universidad Javeriana, Bogotá. franco@javeriana.edu.co y mmmartin@javeriana.edu.co
5Profesor-investigador, Universidad de São Paulo, Instituto de Pesquisas Biomedicas, Brasil. gpadilla@icb.usp.br
Recibido: agosto 21 de 2009 Aprobado: noviembre 23 de 2009
Resumen
Se aislaron diez cepas de actinomicetos nativos del humedal La Conejera a partir de muestras de sedimento y agua, con el fin de determinar la capacidad de resistencia y detoxificación de mercurio, evaluando la presencia del gen mer A involucrado en el proceso. La prueba de resistencia fue realizada mediante un test de sensibilidad tomando concentraciones desde 3,68x10-3 mM hasta 10 mM de HgCl2. Se realizaron curvas de crecimiento de 192 h para las cepas resistentes al mercurio, en un medio suplementado con 0,01 mM y 0,05 mM de HgCl2, realizando una fermentación discontinua y midiendo el crecimiento por la técnica de peso seco. A partir de estos resultados se determinó un tiempo de adaptación de 24 h y una producción máxima de biomasa de 2,72 g·L-1 a la hora 72. Paralelamente, se realizó la secuenciación de los genes de resistencia al mercurio de Streptomyces lividans 1326, por medio del diseño de los oligonucleótidos capaces de amplificar el extremos 3 del gen mer A, codificador de la enzima mercurio reductasa. Se optimizaron las condiciones de amplificación por PCR para obtener un producto amplificado de 686 pb aproximadamente. Finalmente, se realizó una electroforesis en campo pulsado comprobando la presencia de plásmidos en la cepa KH7 con tamaños aproximados de 50, 90 y 300 Kb, no integrados al cromosoma y posiblemente asociados a la resistencia presentada frente al metal.
Palabras clave: Streptomyces, mercurio-resistencia, gen mer A , plásmidos, humedales.
Abstract
Ten native actinomycete strains were isolated from water and sediment samples collected in La Conejera wetlands to assess mercury resistance and detoxification ability for evaluating the presence of the mer A gene involved in that process. Sensitivity assays were performed using 3.68 x 10-3 mM to 10 mM HgCl2 concentrations. Growth curves were generated for each resistant strain after 192 h in discontinuous fermentation in medium supplemented with 0.01 mM and 0.05 mM HgCl2. Biomass growth was then measured by dry weight, maximum value (2.37 gL-1) being obtained after 72 h. Primer pairs were designed based on the sequence encoding the mercury-reductase enzyme in Streptomyces lividans 1326. PCR conditions for amplifying that region were standardised. The mer A gene was identified in the KH7 strain, resulting in an amplified product of around 686 bp. The KH7 strain electrophoresis profile obtained by pulsed field gel electrophoresis showed 50, 90 and 300 Kb plasmid DNA which could be related to metal resistance in this strain.
Key words: Streptomyces, mercury-resistance, mer A gene, plasmid, wetlands.
Introducción
El mercurio es uno de los metales que por su toxicidad representa gran peligro para el medioambiente. Su entrada al medio es causada por procesos naturales o antropogénicos; este último debido al uso indiscriminado de compuestos mercuriales en la industria y la agricultura, así como a la falta de control de afluentes contaminados como sucede en los humedales, lo cual ocasiona daños irreversibles tanto para la biota terrestre como acuática. Los actinomicetos son bacterias Gram positivas que presentan crecimiento filamentoso y alto contenido GC en su ADN; la resistencia de estas bacterias frente a los metales no ha sido investigada con profundidad a pesar de ser metabólica y biosintéticamente versátiles.
Nakahara y et al. (1985) fueron los primeros en estudiar la resistencia frente al mercurio en actinomicetos, la cual fue inducida en Streptomyces lividans 1326, con la posterior clonación y análisis del operón responsable de ésta (Eichenseer y Altenbuchener, 1994). Los genes de resistencia al mercurio en Streptomyces lividans 1326 fueron localizados en un elemento amplificable AUD2, con características semejantes a las de un transposón con secuencias de inserción repetidas al final, debido seguramente a la inestabilidad genética (Eichenseer y Altenbuchner, 1994; Osborn et al., 1997). Streptomyces lividans presenta su resistencia frente al mercurio mediante seis fragmentos abiertos de lectura (ORF) organizados en dos operones de transcripción divergentes. Los genes de regulación y de trasporte forman un operón y las enzimas mercurio reductasa y organomercurio liasa forman el segundo (Brünker et al., 1996).
El gen mer B codifica para la organomercurio liasa la cual rompe los enlaces C-Hg, liberando iones de Hg+2, los cuales se reducen a una forma volátil, el mercurio elemental (Hg0), por la acción de la mercurio reductasa (mer A) (Sedlmer y Altenbuchener, 1992; Rother et al., 1999). Por lo anterior, a partir de los aislamientos de actinomicetos nativos del humedal La Conejera en Bogotá, Colombia, se pretendió evaluar la presencia del genmer A involucrado en la reducción de Hg+2 hasta Hg0.
Materiales y métodos
Muestreo
Diez muestras de sedimento (lodos) y agua, procedentes del humedal La Conejera, en el punto de descarga de los efluentes provenientes de cultivos de flores y de residuos hospitalarios, fueron colectadas a 30 cm de profundidad en recipientes estériles, y transportadas hasta el laboratorio en condiciones de refrigeración. Una vez en el laboratorio fueron procesadas, realizando diluciones seriadas en agua peptonada estéril (0,1% p/v) tomando de cada dilución 0,1 ml para la siembra en superficie sobre placas de agar avena (Thumar y Singh, 2007; Jayasinghe y Parkinson, 2008). Igualmente, fueron recuperadas muestras liofilizadas de actinomicetos nativos aislados del humedal, conservadas en el laboratorio de Microbiología Ambiental y de Suelos de la Pontificia Universidad Javeriana, codificadas como KH1, KH3, KH7, KH16 y AG4, AG5 y AG14.
Ensayos de sensibilidad
El ensayo de sensibilidad fue realizado utilizando el método de anillos de vidrio descrito por Gauze (1965) en placas de agar avena y medio ISP2 (g·L-1: glucosa 4, extracto de malta 10, extracto de levadura 4, agar: 15) (Difco Laboratorios, Detroit, MI, USA) las cuales fueron sembradas e incubadas a 28 °C durante 5 días, teniendo en cuenta como parámetro de selección la formación de halo de menor tamaño alrededor de los anillos. Para este ensayo se utilizaron diferentes concentraciones del metal (10; 5; 3; 2,5; 2; 1,5; 1; 0,5; 0,1 y 0,05 mM) con el fin de determinar la concentración mínima inhibitoria y la máxima tolerable, y de esta forma definir las concentraciones con las cuales se realizarían los siguientes ensayos de la cinética. Se realizaron 5 réplicas por tratamiento.
El crecimiento de las cepas fue comparado usando como control positivo S. lividans 1326 y S. avermitilis cepa sensible al metal (Nakahara et al., 1985), obtenidos del laboratorio de Genética Microbiana del Instituto de Ciencias Biomédicas, Universidad de São Paulo, Brasil.
Cinética de crecimiento
La velocidad de crecimiento de las cepas seleccionadas se determinó mediante la técnica de peso seco a partir de una fermentación discontinua en erlenmeyer de 1000 ml, con 200 ml de medio líquido ISP2 suplementado con mercurio (0,01 y 0,05 mM de HgCl2) en agitación constante (250 rpm) a 28 °C, tomando alícuotas de 10 ml cada 24 h durante 8 días. De esta forma, se determinó el tiempo de adaptación de los microorganismos frente al metal y la cepa con mayor producción de biomasa. Para este ensayo se realizaron 5 réplicas.
Diseño de los oligonucleótidos
Con base en la secuenciación de los genes de resistencia al mercurio de S. lividans 1326 (gen mer A) (Sedlmeier y Altenbuchner, 1992), y con la ayuda del programa Oligos (National Biosciences, Inc, Pymouth MN) se diseñaron unos oligonucleótidos capaces de amplificar la secuencia del gen mer A en las cepas mercurio resistentes. La síntesis de los mismos fue realizada por Invitrogen Life Technologies (São Paulo, Brasil).
Extracción de ADN
Se inocularon las cepas en medio YEME (g·L-1:glucosa 5; extracto de levadura 1,5; bacto pectona 2,5; extracto de malta 1,5; sacarosa 17; MgCl2 (2,5 M) 500 µl; Glicina 20% p/v) que fueron incubadas durante 3 días a 28 °C y 200 rpm. Obtenida la biomasa, se centrifugó el medio para obtener las células, las cuales fueron lavadas con Suc-TE, posteriormente se incubaron con lisozima (5 mg·mL-1) durante 1 h a 37 °C, transcurrido el tiempo se adicionó SDS al 1% p/v (0,7 mL) y EDTA 0,5M (1,2 mL). El producto lisado fue digerido con proteinasa K (1mg·mL-1) y tratado con fenol, fenol-cloroformo (25:24) y solución de cloroformo-alcoholisoamílico (24:1). El ADN fue precipitado con alcohol isopropílico mezclando por inversión (Kieser et al., 2000).
Optimización de las condiciones de la PCR
Las condiciones iniciales para la PCR se basaron en la metodología utilizada por Metsa- Ketela et al. (1999), tomando 10 ng de ADN genómico de S. lividans 1326, 100 pMol de cada primer, 1 mM dNTPS, 2 U Vent Polymerasa (New England Bioabs), 1x Thermopol Buffer (10 mM KCl; 20 mM Tris-HCl pH 8,8; 10 mM (NH4)SO4; 2 mM MgSO4; 0,1% p/v Triton X-100). Manteniendo un volumen total de 50 µl, con ciclos de: 1 ciclo: 96 °C durante 2 min, 56 °C durante 2 min, 73 °C durante 1,5 min; 28 ciclos: 96 °C, 1 min; 56 °C, 2 min; 73 °C, 1,5 min y 1 ciclo: 96 °C, 1 min; 56 °C, 2 min; 73 °C, 8,5 min.
El producto de la PCR fue visualizado en una electroforesis utilizando como tampón de corrida TAE 0,1% p/v (tris base, ácido acético, EDTA pH 8,0) y gel de agarosa al 0,8% p/v. La concentración de ADN para cada muestra fue de 10 µg L-1 respectivamente. El ADN fue separado a 60V durante 180 min.
PFGE (Pulsed-Field gel electrophoresis)
Las cepas resistentes a mercurio fueron incubadas en medio YEME durante 4 días y centrifugadas a 8000 rpm a 5 °C durante 15 min. El precipitado fue suspendido en 5 ml de SucTe (g·L-1: 1,51 Tris-HCl pH 8,0; 4,65 EDTA pH 8,0; 51,35 sacarosa) y diluido hasta obtener una densidad óptica (DO600) de 1,8-2,2 nm de la suspensión celular, con base en la metodología de Kieser et al. (2000) recomendada para bacterias filamentosas. Un volumen de 400 µl de la suspensión bacteriana fue mezclado con 800 µl de la solución 2,0% p/v de agarosa mantenida a 50 °C. Una vez solidificada la solución, los bloques formados fueron transferidos a tubos Falcon con 10 ml de SucTe, y se adicionó lisozima (2mg·mL-1) durante 12 h a 37 °C. Transcurrido el tiempo, los bloques se trataron con DNS durante 24 h a 50 °C. Después de este periodo fueron lavados con tampón TE y tratados con PMFS (40µL·mL-1) dos veces a 4 °C. Finalmente, fueron lavados con tampón TE y mantenidos a 4 °C (Kieser et al., 2000). Las condiciones de corrida fueron de 6s (6,0 v·cm-1 a 14°C) durante 24 h, 70 s (200 v·cm-1 a 14°C) por 15 h y 120 s (200 v·cm-1 a 14°C) durante 11 h con buffer TBE 0,5% v/v (Ravel et al., 1998).
Análisis estadístico
Los resultados obtenidos en las pruebas descritas como parámetro para la selección de las cepas mercurio resistentes fueron analizados con el 95% de confianza, mediante un análisis de varianza bifactorial para posteriormente determinar la homogeneidad entre los grupos según Scheffe, postulando hipótesis nulas donde las diferentes concentraciones de mercurio y las cepas evaluadas producen un mismo halo de sensibilidad (Wayne, 2008).
Resultados y discusión
Diez cepas de actinomicetos fueron aisladas en dos zonas del humedal La Conejera, obteniéndose el mayor número de aislamientos en la zona correspondiente a efluentes procedentes de cultivos de flores. De igual manera, un total de 7 cepas nativas del humedal fueron recuperadas a partir de liofilizados del laboratorio de Microbiología Ambiental y de Suelos de la Pontificia Universidad Javeriana.
Una vez realizado el test de sensibilidad los resultados de las repeticiones se promediaron, dando un solo resultado representativo de las cepas para cada microorganismo enfrentado a las diferentes concentraciones del metal (figura 1). A partir de este resultado las cepas con menor halo de sensibilidad fueron identificadas como KH7 y CR11 respectivamente.
Las cepas evaluadas y las concentraciones propuestas de HgCl2 presentaron diferencias significativas (P=0,05) en la formación de halos de resistencia, parámetro que determinó el rechazo de las hipótesis nulas propuestas en el estudio. La cepa KH7 demostró la mayor resistencia frente al HgCl2 con resultados similares de resistencia comparada con el control positivo S. lividans 1326 (figura 2) tolerando todas las concentraciones propuestas desde 0,05 a 10 mM con halos inferiores a 6 mm. Los resultados anteriores proponen la relación entre la resistencia al HgCl2 y la actividad de la enzima mercurio reductasa codificada por los genes mer A. La cepa KH7 muestra una fase logarítmica hasta la hora 72 seguida de la fase estacionaria hasta la hora 120; la curva control presenta un comportamiento similar con una fase logarítmica hasta la hora 120 y producción máxima de biomasa de 2,93 g·L-1. Hay similitudes con la curva control, sin afectar la actividad enzimática y el crecimiento del microorganismo (figura 3). A una concentración de 0,05 mM de HgCl2 se presenta una marcada disminución de la biomasa celular (0,73 g·L-1) determinada por el efecto tóxico del HgCl-, al compararla con la concentración de 0,01 mM donde se obtuvo como valor máximo de producción de biomasa 3,10 g·L-1 en la hora 96. Esto se puede explicar por la formación de enlaces covalentes con los grupos sulfhidrilo de la cisteína presentes en las proteínas de la pared y membrana celular, siendo posiblemente la causa de la baja cinética registrada.
En relación con la secuenciación del gen de resistencia al mercurio de Streptomyces lividans 1326 (gen mer A), a partir del cual se diseñaron los oligonucleótidos capaces de amplificar los extremos 3 del gen mer A, permitió que se cumplieran las variables propuestas para el éxito en la amplificación. Los oligonucleótidos construidos correspondieron al oligonucleótido forward secuencia (5 a 3): GAC CTC CAC CAC CGC CAT G. La secuencia del oligonucleótido reverso fue (5 a 3): GGG ACA GGG TGC GGG TCT G. Las condiciones de corrida para la PCR fueron optimizadas y estandarizadas para la amplificación del gen mer A encargado de la síntesis de la enzima mercurio reductasa.
Con una disminución de la temperatura de anillaje de 56 °C a 50 °C y utilizando una temperatura de extensión para cada ciclo de 68 °C, se logró obtener un fragmento amplificado de aproximadamente 686 pb (figura 4) correspondiente al gen mer A manteniéndose el resto del programa igual al establecido previamente.
Con la realización de la PFGE fue determinada la presencia de tres plásmidos naturales lineales, detectados en la cepa KH7 con un tamaño aproximado de 50, 90 y 300 Kb no integrados al cromosoma (figura 5). La migración de los plásmidos fue determinada de acuerdo con los diferentes pulsos de corrida establecidos en la metodología, lo cual indicó que los tres plásmidos son lineales y no circulares al poseer proteínas ligadas covalentemente a las extremidades 5 del ADN. La migración de los plásmidos fue posible solo al tratar el ADN con proteinasa K, usada después de causar la lisis celular, para ayudar a liberar el ADN de los restos de célula y probablemente eliminar los enlaces proteicos ligados a los plásmidos (Ravel et al., 1998). Con relación al ADN de S. lividans 1326, fue imposible obtener fragmentos claros asociados a los plásmidos, posiblemente derivados de la susceptibilidad que se puede presentar frente a la degradación del ADN durante la corrida de la PFGE.
En los actinomicetos, la síntesis de la enzima mercurio reductasa se asocia con la presencia de un plásmido lineal gigante pbd2 en Rhodococcus erythropolis (Dabrock, 1994; Huddleston et al., 2006) y los prj3h, pRJ3L y prj28 encontrados en especies relacionadas con S. pseudogriseolus y S. tendae (Ravel et al., 1998) donde se ha demostrado la presencia de genes mer involucrados en la remoción del mercurio que codifican para enzimas intracelulares como la Hg reductasa y la organomercurioliasa. El Hg+2 es ligado a grupos sulfhidrilo de la cisteína adyacente al sitio activo de la enzima reductasa, reduciendo dicho ión a Hg0, siendo menos tóxico y difusible al exterior de la célula (Williams, 1997).
Aunque es común encontrar estos plásmidos lineales gigantes en las especies de Streptomyces, muchas de sus funciones son desconocidas hasta ahora. Diversos plásmidos lineares se han asociado a fenotipos específicos, incluyendo sistemas de restricción y modificación (Zotchev y Schrempf, 1993), producción de antibióticos, resistencia a metales pesados y habilidad en la trasformación de productos xenobióticos (Meinhardt et al., 1997; Mochizuki et al., 2003).
Conclusión
En conclusión, la actividad metabólica de la cepa KH7, permite la reducción del Hg+2 a mercurio Hgo, al presentar en su genoma genes encargados de la función codificadora para la enzima mercurio reductasa. Lo anterior se vio reflejado en que bajas concentraciones de HgCl2 (0,01 mM) no alteraron el adecuado desarrollo reflejado en la cinética de crecimiento de los actinomicetos mercurio-resistentes, y en que se consiguió la amplificación de un fragmento de 686 pb correspondiente al extremo 3´ del merA involucrado en la reducción del mercurio. De esta manera, se permite inferir que la diversidad metabólica de estos microorganismos hace de ellos candidatos importantes para su uso en biorremediación.
Agradecimientos
A la Pontificia Universidad Javeriana por la financiación del proyecto, a la Universidad de São Paulo, Instituto de Ciencia Biomédicas en Brasil por la aceptación de la pasantía y asesoría en Biología Molecular.
Referencias bibliográficas
1 Amoroso, M. J., Castro, G. R., Carlino, F. J., Romero, N. C., Hill, R. T., Oliver, G. 1998. Screening of heavy metaltolerant actinomycetes isolated from the Salí River. Journal of General and Applied Microbiology 44: 129- 132.
2 Brünker, P., Rother, D., Sedlmeier, R., Klein, J., Mattes, R., Altenbuchner, J., Sedlmeier, R. 1996. Regulation of the operon responsible for broad-spectrum mercury resistance in Streptomyces lividans 1326. Molecular and General Genetics 251: 307-15.
3 Dadrock, B. 1994. Identification and characterization of a transmissible linear plasmid from Rhodococcus erythopolis BD2 that encodes isopropylbenzene and trichloroethane catabolism. Applied and Environmental Microbiology 60: 853-860.
4 Einchenseer, C., Altenbuchner, B. 1994. The very large amplifiable element AUD2 from Streptomyces lividans 66 has insertion sequence-like repeat at its ends. Journal of Bacteriology 176: 7107-12.
5 Gauze, G. F. 1965. Problems in the classification of antagonist of Actinomycetes. La Habana, Cuba: Ed. Medriz- Mosco, p. 22.
6 Huddleston, J. R., Zak, J. C., Jeter, R. M. 2006. Antimicrobial Susceptibilities of Aeromonas spp. Isolated from Environmental Sources. Applied and Environmental Microbiology 72: 7036-7042.
7 Jayasinghe, D. B. A. T., Parkinson, D. 2008. Actinomycetes as antagonists of litter decomposer fungi. Applied and Soil Ecology 38: 109-118.
8 Kieser, T., Bibb, M. J., Buttner, M. J., Chater, K. F., Hopwood, D. A. 2000. Practical Streptomyces genetics. United Kingdom: John Innes Foundation, Norwich. pp. 161-211.
9 Meinhardt, F., Schaffrath, R., Larsen, M. 1997. Microbial linear plasmids. Applied Microbiology and Biotechnology 47: 329-336.
10 Metsa-Ketela, M., Salo, V., Halo, L., Hautala, J., Hakala, J., Mantsala, P., Ylihonko, K. 1999. An efficient approach for screening minimal PKS genes from Streptomyces. FEMS Microbiology Letters 180: 1-6.
11 Mochizuki, S., Hiratsu, K., Suwa, M., Ishii, T., Sugino, F., Yamada, K., Kinashi, H. 2003. The large linear plasmid pSLA2-L of Streptomyces rochei has an unusually condensed gene organization for secondary metabolism. Molecular Microbiology 48: 1501-1510.
12 Nakahara, H., Schottel, J., Yamada, T., Mikakawa, Y., Asakawa, M., Harville, J., Silver, S. 1985. Mercuric reductase enzymes from Streptomyces species and group B Streptococcus. Journal of General Microbiology 132: 1053-1059.
13 Osborn, A. M., Bruce, K. D., Strike, P., Ritchie, D. A. 1997. Distribution, diversity and evolution of the bacterial mercury resistance (mer) operon. FEMS Microbiology Reviews 19: 239-262.
14 Ravel, J., Schrempf, H., Hill, R. 1998. Mercury resistance is encoded by transferable giant linear plasmids in two Chesapeake Bay Streptomyces strains. Applied and Environmental Microbiology 64 (9): 3383-3388.
15 Rother, D., Mattes, R., Altenbuchner, J. 1999. Purification and characterization of MerR, the regulator of the broad-spectrum mercury resistance genes in Streptomyces lividans 1326. Molecular and General Genetics 262: 154-162.
16 Sedlmeier, R., Altenbuchner, J. 1992. Cloning and DNA sequence analysis of the mercury resistance genes ofStreptomyces lividans. Molecular and General Genetics 236: 76-85.
17 Silver, S., Misra, T. K. 1998. Plasmid-Mediated Heavy Metal resistences. Applied and Environmental Microbiology 42: 717-743.
18 Thumar, J., Singh, S. P. 2007. Two-step purification of a highly thermostable alkaline protease from salt-tolerant alkaliphilic Streptomyces clavuligerus strain Mit-1. Journal of Chromatography B 854:198-203.
19 Wayne, D. 2008. Bioestadística: base para el análisis de las ciencias de la salud. 5 ed. México: Limusa. pp. 345- 446.
20 Williams, J. 1997. Bacterial resistance and detoxification of heavy metals. Enzyme and Microbial Technology 18 (6): 85-90.
21 Zotchev, S., Schrempf, C. 1993. The linear Streptomyces plasmid pbl1: analyses of transfer functions. Molecular and General Genetics 242: 374-38.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2009 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).