Diseño de casetes de expresión que confieran tolerancia a sequía y a glufosinato en maíz (Zea mays)
Design of expression cassettes that confer drought and glufosinate tolerance to maize (Zea mays)
DOI:
https://doi.org/10.15446/abc.v21n3.51170Palabras clave:
estrés abiótico, tolerancia a herbicida, transgénesis, uso codónico. (es)abiotic stress, codon usage, herbicide tolerance, transgenesis. (en)
Descargas
Como primera aproximación en la obtención de una línea transgénica de maíz tolerante a sequía y al herbicida glufosinato de amonio, se seleccionaron genes y elementos reguladores para el diseño in silico de casetes de expresión, a través del análisis de literatura científica y bases de datos de genes y patentes. Las secuencias génicas fueron modificadas con base en el criterio de uso codónico del maíz para optimizar su expresión. Los casetes de expresión diseñados con el software DNA 2.0., fueron sintetizados por una empresa especializada. La presencia del transgen y la expresión a nivel de mARN fue demostrada mediante PCR y RT-PCR en la planta modelo Nicotiana benthamiana transformada vía Agrobacterium tumefaciens. Un ensayo preliminar in vitro en condiciones simuladas de sequía en medio MS con PEG (PM 6000)10 % no demostró incremento notorio en la tolerancia de las plántulas transformantes, posiblemente debido a que el uso codónico del diseño no favorece la expresión génica en la planta modelo.
As a first approach in obtaining a transgenic drought and glufosinate ammonium tolerant maize line, genes and regulatory elements for the in silico design of the expression cassettes were selected through analysis of scientific literature and databases of genes and patents. Gene sequences were modified based on the criterion of maize codon usage to optimize their expression. The constructs designed with DNA 2.0 Software, were synthesized by a specialized company. The presence of the transgene and the expression of mRNA was demonstrated by PCR and RT-PCR in the model plant Nicotiana benthamiana transformed via Agrobacterium tumefaciens. A preliminary experiment in vitro under simulated drought conditions in MS medium with 10 % PEG (PM 6000) showed no noticeable increase in drought tolerance of the transformants, possibly because the codon usage of the design does not promote gene expression in the model plant.
DOI: https://doi.org/10.15446/abc.v21n3.51170
DISEÑO DE CASETES DE EXPRESIÓN QUE CONFIERAN TOLERANCIA A SEQUÍA Y A GLUFOSINATO EN MAÍZ (Zea mays)
Design of Expression Cassettes that Confer Drought and Glufosinate Tolerance to Maize (Zea mays)
Andrea CARREÑO-VENEGAS1; Alejandro CHAPARRO-GIRALDO2.
1,2 Grupo de Ingeniería Genética de Plantas. Departamento de Biología & Instituto de Genética, Universidad Nacional de Colombia, Sede Bogotá. Cra. 30 n°. 45-03, edificio 421, oficina 208. Bogotá, Colombia.
For correspondence. afcarreno@unal,edu.co; achaparrog@unal.edu.co
Received: 1st July 2015, Returned for revision: 7th April 2016, Accepted: 30 June 2016.
Associate Editor: Adriana Almeida.
Citation/Citar este artículo como: Carreño-Venegas A, Chaparro-Giraldo A. Diseño de casetes de expresión que confieran tolerancia a sequía y a glufosinato en maíz (Zea mays). Acta biol. Colomb. 2016;21(3):555-570. DOI: https://doi.org/10.15446/abc.v21n3.51170
RESUMEN
Como primera aproximación en la obtención de una línea transgénica de maíz tolerante a sequía y al herbicida glufosinato de amonio, se seleccionaron genes y elementos reguladores para el diseño in silico de casetes de expresión, a través del análisis de literatura científica y bases de datos de genes y patentes. Las secuencias génicas fueron modificadas con base en el criterio de uso codónico del maíz para optimizar su expresión. Los casetes de expresión diseñados con el software DNA 2.0., fueron sintetizados por una empresa especializada. La presencia del transgen y la expresión a nivel de mARN fue demostrada mediante PCR y RT-PCR en la planta modelo Nicotiana benthamiana transformada vía Agrobacterium tumefaciens. Un ensayo preliminar in vitro en condiciones simuladas de sequía en medio MS con PEG (PM 6000)10 % no demostró incremento notorio en la tolerancia de las plántulas transformantes, posiblemente debido a que el uso codónico del diseño no favorece la expresión génica en la planta modelo.
Palabras clave: estrés abiótico, tolerancia a herbicida, transgénesis, uso codónico.
ABSTRACT
As a first approach in obtaining a transgenic drought and glufosinate ammonium tolerant maize line, genes and regulatory elements for the in silico design of the expression cassettes were selected through analysis of scientific literature and databases of genes and patents. Gene sequences were modified based on the criterion of maize codon usage to optimize their expression. The constructs designed with DNA 2.0 Software, were synthesized by a specialized company. The presence of the transgene and the expression of mRNA was demonstrated by PCR and RT-PCR in the model plant Nicotiana benthamiana transformed via Agrobacterium tumefaciens. A preliminary experiment in vitro under simulated drought conditions in MS medium with 10 % PEG (PM 6000) showed no noticeable increase in drought tolerance of the transformants, possibly because the codon usage of the design does not promote gene expression in the model plant.
Keywords: abiotic stress, codon usage, herbicide tolerance, transgenesis.
INTRODUCCIÓN
El cambio climático es un fenómeno global que ha causado pérdidas económicas en la agricultura, ya que disminuye drásticamente el rendimiento de los cultivos. El Panel Intergubernamental sobre el Cambio Climático (Change, 2001) plantea que las zonas agrícolas de mayor riesgo a causa del cambio climático son las sub-tropicales y tropicales. En relación con las áreas agrícolas más secas, se esperan fenómenos de salinización y desertificación causando disminuciones importantes en la productividad de cultivos y ganadería, poniendo en riesgo la seguridad alimentaria (Change, 2001).
El estrés por sequía en plantas, se puede definir como una situación ambiental, en la cual el recurso hídrico es escaso, por lo que la tasa de transpiración excede la toma de agua, llevando a la reducción del potencial hídrico y de la turgencia de la planta; interfiriendo con las funciones normales y desencadenando respuestas fisiológicas, bioquímicas y moleculares específicas (Schachtman y Goodger, 2008; Shao et al., 2008; Edgerton, 2009; Ashraf, 2010).
El cultivo de maíz (Zea mays) es muy sensible al estrés por déficit de hídrico (Boyer y Westgate, 2004), especialmente las etapas de polinización y desarrollo del embrión se ven muy afectadas por el suministro de agua del suelo (Grant et al., 1989; Bolaños y Edmeades, 1996). La sequía en estas etapas del cultivo tiene un efecto multiplicador sobre el rendimiento, al reducir la formación de reservas causada por dificultades en la polinización o por la detención del crecimiento de óvulos fertilizados (Boyer y Westgate, 1994; Lafitte, 2001). Cuando el maíz florece bajo sequía, se presenta un retraso en la floración femenina o en la emisión de estigmas y un incremento en el periodo entre la floración masculina y femenina, dando lugar al intervalo de antesis y emisión de estigmas (Ngugi et al., 2013).
Los principales productores del grano de maíz son Estados Unidos, China y Brasil con una producción de 273,832, 205,600 y 81,000 toneladas métricas para el 2013, que representan cerca del 37 %, 28 % y 11 % de la producción mundial respectivamente (Statista, 2015). La caída en la producción nacional de los Estados Unidos podría impactar la producción mundial. Para el año 2012, en gran parte del medio oeste de este país, la sequía fue responsable de la reducción de un 15 % en la producción y un 21 % en el rendimiento nacional de maíz, en comparación con los niveles medios de los años 2009 al 2011 (Edmeades, 2013).
La ingeniería genética, que permite la introducción de genes específicos, restringiendo la transferencia de genes no deseados del organismo donador, hace posible que un mismo organismo cuente con diferentes características deseadas a través de la piramidación de genes (Gosal et al., 2009). Es una herramienta valiosa para la introducción de características deseables en plantas, que son difíciles de lograr mediante técnicas convencionales, como es el caso de la tolerancia a sequía, debido a la baja heredabilidad del carácter (Long y Ort, 2010), o a que no se encuentran naturalmente en estos organismos.
Fueron seleccionados tres genes denominados TaDREB3 AP2 (Lopato et al., 2006; Morran et al., 2011), AtZAT10 (Meissner y Michael,1997; Mittler et al., 2006) y CspB (Willimsky et al., 1992; Castiglioni et al., 2008), que pueden conferir tolerancia a sequía.
Todos los DREB/CBFs pertenecen a la superfamilia de factores de trascripción (FTs) AP2/ERF, que es especifica de plantas, y que son considerados los principales FTs involucrados en la regulación de la expresión de genes inducidos por deshidratación y frío. Sin embargo también cumplen papeles importantes en el desarrollo de semillas, hojas y flores (Long y Ort, 2010; Yang et al., 2010,).DREB/CBF hacen parte de los FTs que responden a deshidratación, pero no al ácido abscísico (ABA). Se unen específicamente a elementos DRE, identificados por primera vez en el promotor del gen RD29A de Arabidopsis (Yamaguchi-Shinosaki y Shinosaki, 1994). Seis factores de transcripción DREB que incluyen cuatro genes DREB1/CBF y dos genes DREB2 fueron identificados en Arabidopsis. La expresión de DREB1/CBFs es inducida por sequía, sal y frio, mientras que la expresión de DREB2 es únicamente inducida por sequía y sal (Liu et al., 1998; Sakuma et al., 2006b; Morran et al., 2011). Diferentes estudios han informado mejora en la tolerancia a sequía mediante transformación genética en plantas cultivadas con tales factores de transcripción, en tomate (Solanum lycopersicum) (Hsieh et al., 2002), trigo (Triticum aestivum) (Pellegrineschi et al., 2004; XiaoYan et al., 2005; Wang et al., 2006), maíz (Al-Abed et al., 2007; Zhang et al., 2010) y arroz (Oriza sativa) (Oh et al., 2005; Oh et al., 2007; Chen et al., 2008; Xiao et al., 2009); sin embargo, uno de los principales inconvenientes en transformación genética de plantas con DREB/CBF han sido los defectos en el crecimiento cuando se expresa constitutivamente, lo cual se ha minimizado mediante el uso de promotores inducibles por estrés.
El gene ZAT10, forma parte de un grupo de supresores transcripcionales de la familia génica C2H2 de dedos de zinc que se caracteriza por contener un dominio de represión amfifílico (EAR) asociado-ERF, involucrado en la modulación la respuesta de defensa de las plantas al estrés abiótico a través de la regulación transcripcional de diferentes genes reporteros y de defensa (Meissner y Michael, 1997; Ohta et al., 2001; Englbrecht et al., 2004; Sakamoto et al., 2004; Ciftci-Yilmaz y Mittler, 2008). ZAT10 puede unirse al promotor de RD29A (un gen clásico de respuesta a estrés) y reprimir su transcripción (Sakamoto et al., 2000; Lee et al., 2002). Se ha asociado estrechamente con respuesta a estrés oxidativo en plantas (Rizhsky et al., 2004; Davletova et al., 2005; Mittler et al., 2006) y se sugiere que juega un papel clave en regulación positiva y negativa de la defensa en plantas a determinados estreses abióticos. Su papel principal involucra la modulación de la respuesta a especies reactivas de oxigeno (ROS, de sus siglas en ingles), ya sea a través de la activación directa de la transcripción de genes de respuestas de ROS a través de la supresión de represores de estos genes incrementando la tolerancia de las plantas a estrés abiótico (Mittler et al., 2006). La expresión constitutiva de la proteína ZAT10 C2H2-EAR en Arabidopsis incrementa la expresión de FSD1 (ferro-superoxido dismutasa) y APX2 (ascorbato peroxidasa), pero no parece estar involucrada con la expresión de otros transcritos de respuesta a sal, frío o sequía. Adicionalmente confiere mayor tolerancia a salinidad, calor y estrés osmótico (Mittler et al., 2006).
Genes que codifican para proteínas que actúan como chaperonas moleculares tipo heat-shock protein de origen vegetal y cold shock protein (Csps) de origen bacteriano (Escherichia coli y Bacillus subtilis), han permitido mayor tolerancia a estrés por sequía tanto en plantas cultivadas como en plantas modelo, mediante ingeniería genética. Las Csps son una familia de proteínas pequeñas, altamente conservadas en su estructura, con masa molecular de aproximadamente 7,4 kDa. Son capaces de unirse a una sola cadena del ADN o ARN, a través de los motivos de unión a ácidos nucleicos RNP1 y RNP2. La unidad estructural característica de estas proteínas es el dominio cold shock (CSD) que puede ser hallado en otras proteínas involucradas en regulación transcripcional que se encuentran en una variedad de organismos, desde bacterias a vertebrados (proteínas Y-box) (Horn et al., 2007). CspA y CspB bacterianas han sido expresadas en Arabidopsis, arroz (Oriza sativa) y maíz (Zea mays), confiriendo incremento en la tolerancia a sequía, frío y calor a través de la protección y el favorecimiento del crecimiento vegetativo, fotosíntesis, y desarrollo reproductivo (Castiglioni et al., 2008). El gen CspB es el único hasta el momento, con el que se ha logrado el primer cultivo comercial de maíz tolerante a sequía (Droughtgard™ con el evento MON87460). Su papel en la tolerancia en plantas se atribuye principalmente a su actividad como chaperona RNA (Edmeades, 2013; James, 2014). Tal actividad difiere de lo observado con otras proteínas chaperonas que usualmente ayudan a formar correctamente estructuras secundarias y terciarias después de desestabilizar las estructuras secundarias equivocadas, puesto que al parecer las Csps sólo destruyen estructuras secundarias no deseadas (Horn et al., 2007).
La variación en el manejo de la densidad de las plantas y control de malezas, son prácticas agrícolas, que además de la irrigación, pueden alterar substancialmente la cantidad de agua capturada por la planta, determinando la disponibilidad de agua para la transpiración (Edmeades, 2013), dicha disponibilidad podría favorecerse en cultivos tolerantes a herbicidas. La tolerancia a glufosinato de amonio en plantas se ha conseguido por medio de ingeniería genética, transfiriendo el gen bacteriano pat o bar, que codifican la enzima fosfinotricina N-acetil-transferasa (PAT), capaz de inactivar el ingrediente activo del glufosinato de amonio (fosfinotricina), que en plantas no transformadas, bloquea la enzima glutamina sintetasa, involucrada en el metabolismo del nitrógeno y en la detoxificación de amonio, llevando a la planta a la muerte por intoxicación debido a la acumulación de niveles letales de amonio y deficiencia en la síntesis proteínas (De Block et al., 1987).
En el presente estudio, se diseñaron in silico tres casetes de expresión que contienen un gen que pueden conferir tolerancia a sequía y el gen bar que confiere tolerancia a glufosinato de amonio. La integración de los casetes expresión y la presencia de los transcritos correspondientes (mRNA) a las regiones codificantes fueron evaluados en planta modelo N. Benthamiana, transformada vía A. tumefaciens. Los resultados de este estudio, son preliminares en el desarrollo y obtención futura de líneas de maíz comerciales con dichas características.
MATERIALES Y MÉTODOS
Búsqueda y selección de genes de tolerancia a sequía y a glufosinato de amonio
Los genes usados en el diseño de los casetes de expresión, fueron seleccionados a partir de literatura científica con base en la pertinencia de los estudios y los resultados obtenidos. Las secuencias nucleotídicas y aminoacídicas, fueron obtenidas a partir de la base de datos de patentes The Lens (https://www.lens.org/lens/) y/o el Centro Nacional de Información sobre Biotecnología de los Estados Unidos (NCBI) (http://www.ncbi.nlm.nih.gov/gene).
Se revisaron 105 artículos científicos de transgénesis sobre tolerancia a sequía en plantas modelo y plantas cultivadas, incluido el maíz (Zea mays). Estos estudios fueron consultados en la base de datos de la Universidad Nacional de Colombia, principalmente en: Academic Search Complete, Science Direct, Springer Journal, Journal Storage y Nature.com, entre el periodo 1995 a 2013. En general se tuvo en cuenta el papel del gen en la tolerancia a sequía y los parámetros fisiológicos disponibles con los que se evaluó la tolerancia: capacidad germinativa en condiciones de sequía o condiciones similares (en suelo o in vitro), daño celular, actividad fotosintética, potencial osmótico, fenología y rendimiento.
Los genes seleccionados de tolerancia a sequía para el diseño de los casetes de expresión fueron TaDREB3 AP2 (Lopato et al., 2006) AtZAT10 (Meissner y Michael,1997) y CspB (Willimsky et al., 1992), que provienen de trigo (Triticum aestivum), Arabidopsis thaliana, y Bacillus subtilis, respectivamente. Teniendo en cuenta como criterios de selección: i) fuerte evidencia empírica sobre tolerancia a sequía, de acuerdo a parámetros fisiológicos evaluados en ensayos in vitro, invernadero o campo; ii) valores de rendimiento en campo para el caso de las plantas cultivadas; iii) ausencia de efectos pleiotrópicos no deseados o productos génicos que actúen en partes altas de las vías de señalización de estrés; y iv) estudios más recientes. Por otra parte, el gen bar fue elegido debido a que se ha usado como marcador de selección en medio de cultivo, se encuentra en cultivos comerciales tolerantes a glufosinato y se ha probado en maíz (patente US6395966B1).
Diseño y síntesis de casetes de expresión
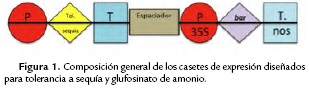
Los componentes básicos de los casetes de expresión comprenden una secuencia promotora, un marco de lectura abierto (de las siglas en ingles ORF), y una región terminadora no traducida 3' que en eucariotas, generalmente contiene un sitio de poliadenilación (Cai et al., 2007). Cada uno de los tres casetes de expresión diseñados (CspB+bar, TaDREB3+bar, AtZAT10+bar), contiene dos constructos, uno que confiere tolerancia a sequía y otro a glufosinato de amonio con sus respectiva región promotora y terminadora, además una región no codificante ubicada en el medio (Fig. 1).
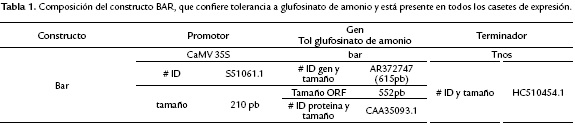
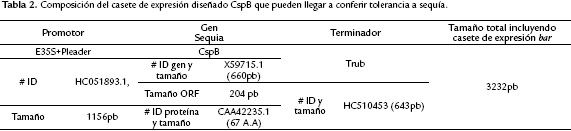
Cada una de las secuencias génicas se diseñaron, dirigidas por un promotor constitutivo de la siguiente manera: i) CspB, dirigido por CaMV35s potenciado, y una secuencia líder se petunia (E35S+Pleader), ii) AtZAT10, dirigido por la región promotora de la Ubiquitina de maíz Ubi-1 y iii) bar, dirigido por CaMV 35S. El gen TaDREB3 fue dirigido por el promotor inducible por sequía rab17. La región terminadora de CspB y TaDREB3 fue el terminador de la RuBisCo (Trub), mientras que la región terminadora de AtZAT10 y Bar fue el de nopalina sintasa (Tnos). En todos los casos, para separar el constructo del gen que confiere tolerancia a sequía del constructo del gen bar, se introdujo una región no codificante (intrón) de cloroplasto de Acer pseudoplatanus (Taberlet et al., 1991), que cumple el papel de espaciador. A continuación se muestra la composición detallada de cada uno de los constructos: el número de identificación (# ID) en el GenBank de las secuencias nucleotídicas (promotoras-codificantes-terminadoras) y aminoacídicas, el tamaño en pares de bases (completa/ORF) y aminoácidos, respectivamente (Tab. 1, 2, 3 y 4).
El diseño in silico se hizo con el programa Gen Designer 2.0, de uso libre (Villalobos et al., 2006; DNA 2.0, Inc.). El diseño incluyó: i) Obtención del marco de lectura abierto (ORF) que fue comprobado a través de la herramienta "ORF finder" del NCBI; ii) Comparación de la secuencia de aminoácidos que codifica la secuencia ORF diseñada versus la secuencia de aminoácidos correspondiente, obtenida en el NCBI o en la patente (EMBL-EBI, 2015); iii) Optimización de la región codificante de acuerdo al uso codónico de Zea mays, bajo la opción "DNA/ Guide Random (Puigbo et al., 2007; OPTIMIZER, 2007); iv) Adición de sitios de restricción dentro de los casetes de expresión, que permitan aislar secuencias de interés del casete o hacer mapas de restricción; v) reconfirmación del ORF, posterior a la adición de los sitios de clonación y vi) reconfirmación de la identidad usando la herramienta Blastx (BLAST®). Las secuencias diseñadas mediante bioinformática, fueron sintetizadas químicamente y transferidas al vector de clonación pCAMBIA 1301 por la empresa GENEray Biotechnology en Shangai, China.
Transformación y caracterización fenotípica/ molecular de cepas recombinantes
La transformación de las cepas bacterianas comerciales DH5Ade E. coli (One shot® TOP10 Electrocomp TM E. coli, de invitrogen), y LBA4404 de A. tumefaciens, se realizó a través de electroporación, con el propósito de clonar cada uno de los casetes de expresión y aislar suficiente ADN plasmídico. Se almacenaron muestras de cepas recombinantes de E. coli y A. tumefaciens a -80 °C, para los ensayos de transformación genética de plantas. Las cepas bacterianas posiblemente transformadas, fueron caracterizadas fenotípicamente. Escherichia coli, fue cultivada por agotamiento en medio de LB sólido con el antibiótico de selección kanamicina, teniendo en cuenta que plásmido pCAMBIA 1301 contiene un gen de resistencia a este antibiótico, mientras que A. tumefaciens fue seleccionado con kanamicina y estreptomicina (0,05 mg/ml), el uso del segundo antibiótico se estableció teniendo en cuenta la resistencia cromosómica que expresa la bacteria.
La caracterización molecular se llevó a cabo mediante PCR de colonias utilizando pares de cebadores diseñados con el programa Primer3Plus (Rozen y Skaletsky, 2000) de acuerdo a las secuencias nucleotídicas de cada gen. CspB cuyo amplicón esperado es de 152 pb (5'-GGGAAGGTCAAGTGGTTCAA-3' y 5'-CCCTCCACAATCTCAAAGGA-3'), TaDREB3 cuyo amplicón esperado es de 181 pb (5'-GACCCCGCCACTTTAAGAAC-3' y 5'-CCCCGTACATTGCGTAAGAA-3'), AtZAT10 cuyo amplicón esperado es de 212 pb (5'-TCGTACAAGTGCTCCGTTTG-3' y 5'-TGACCGCTAGGGAAACTCTT-3') y bar (5'-TTTGCACCA TCGTCAATCAT-3' y 5'-TCGTGCATCCTCACACTAGG-3') cuyo amplicón esperado es de 352 pb. Las colonias que fueron positivas para PCR, se subcultivaron en medio LB y el antibiótico de selección respectivo.
Transformación de plantas
Plantas de tabaco (Nicotiana benthamiana) transformadas independientemente con los casetes de expresión CspB, TaDREB3 y AtZAT10, a través de A. tumefaciens, de acuerdo con un protocolo básico de transformación que involucra seis pasos principales: i) densidad óptica (DO a 600 nm) de la suspensión bacteriana de A. tumefaciens entre 0.05-0.1; ii) explantes de hoja de 1 cm x 1cm aproximadamente (50 o más explantes por ensayo), sumergidos por dos minutos en el cultivo bacteriano agitando suave y constantemente a 30 rpm; iii) secado y siembra de los explantes en medio de cocultivo ( Sales Murashige-Skoog (1962) + Bencilaminopurina (BAP) 1 mg/l + acetociringona 200 uM), durante 24 horas en oscuridad en cuarto de crecimiento (70 % de humedad relativa (HR) y 28 °C); iv) lavados de los explantes para eliminar A. tumefaciens con antibiótico; v) siembra de explantes en medio de selección (MS + BAP + carbenicilina 500 mg/l + Fosfinotricina (PPT) 0,8 mg/l (Jimenéz, 2014) o 2 mg/l (De Block et al., 1987) durante cuatro semanas aproximadamente, con un cambio de medio a la segunda semana; y vi) siembra de brotes en medio de elongación (MS); el medio MS se compone de sales MS 4g/l, vitamina Gamborg B-5 1ml/l, sacarosa 30 g/l y gelzan (Gelrite®) 2,5g/l, como agente gelificante.
Tanto la selección como la elongación se llevaron a cabo en cuarto de crecimiento (70 % HR, 28 °C, 16/8 fotoperiodo). Todos los ensayos de transformación contaron con controles de explantes no cocultivados con la bacteria (no transformados) en medio de regeneración sin selección y en medio de regeneración con selección; para un total de 25 explantes control en medio de selección y en medio sin selección, respectivamente. Se realizaron algunas modificaciones al protocolo base, debido a los altos niveles de contaminación por la bacteria. Respecto a los lavados de los explantes para la eliminación de A. tumefaciens, el protocolo inicial sugería tres lavados por 60 minutos cada uno en medio MS liquido con o sin antibiótico; el primero en medio MS, el segundo y tercero en MS + cefotaxime (500mg/ml) y el tercero igual al anterior. Se modificó a cinco lavados consecutivos de diez minutos con MS, 30 minutos con MS, dos lavados de 60 minutos cada uno con MS + cefotaxime 500 mg/l y 30 minutos con MS + cefotaxime 500 mg/l, tales modificaciones permitieron solucionar el problema de contaminación por A. tumefaciens. Otra modificación fue el uso de carbenicilina en vez de cefotaxime en el medio de elongación (Horsch et al., 1985; Silva y Fukai, 2001).
Caracterización molecular de N. benthamiana mediante PCR y RT-PCR
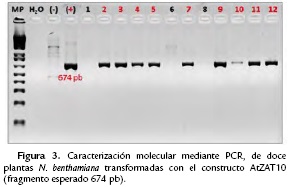
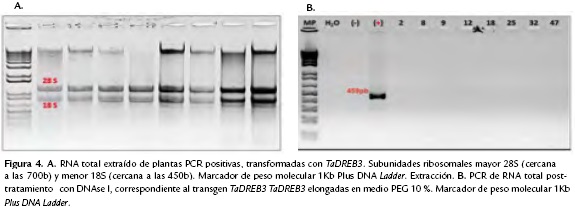
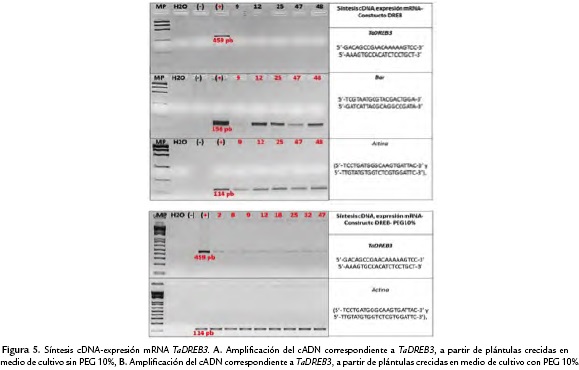
Plántulas regeneradas en medios de selección con PPT a partir de ensayos de transformación independientes con los casetes de expresión correspondientes a CspB, TaDRE3B y AtZAT10, fueron evaluadas mediante PCR con el kit KAPA3G (KAPABIOSYSTEMS) que permite realizar la PCR directamente con el tejido de hoja, con lo que se determinó la presencia o ausencia del transgen. Nuevos cebadores fueron diseñados con el programa Primer3Plus (Rozen y Skaletsky, 2000) de acuerdo a las secuencias nucleotídicas de cada gen. CspB cuyo amplicón esperado es de 200 pb (5'-ATGCTCGAAGGGAAGGTC-3' y 5'-GCCTCCTTTGTCACATTAGCA-3'), TaDREB3 cuyo amplicón esperado es de 459 pb (5'-GACAGCCGAACAAAAAGTCC-3' y 5'-AAAGTGCCACATCTCCTGCT-3'), AtZAT10 cuyo amplicón esperado es de 674 pb (5'-GGCGCTAGAGGCTCTGACTA-3' y 5'-GCAGTTTCACTGGGAAGTCG-3') y bar (5'-TCGTAATGCGTACGACTGGA-3' y 5'-GATCATTA GGCAGGCCGATA-3') cuyo amplicón esperado de 158 pb. De las plántulas caracterizadas como PCR positivas, se eligió por casete de expresión, al azar entre cinco a siete, de acuerdo con el material disponible. Con el fin de comprobar o no la expresión a nivel de mARN, a dichas plántulas se les realizó RT-PCR. Hojas de plantas elongadas de tres semanas de edad contados a partir del paso de los brotes al medio de elongación fueron utilizadas para la extracción de ARN mediante el kit de NORGEN BIOTEK-CORP. El ARN fue sometido a tratamiento con DNAse I, RNase-free (Termo Scientific) y posteriormente se comprobó mediante PCR la ausencia de ADN en las muestras. A continuación se sintetizó cADN con el kit First Strand cDNA Synthesis (Termo Scientific) y a partir de este, se realizaron las PCRs correspondientes. Los primers utilizados en la PCR de ARN tratado con DNAse I y cADNs son los mencionados anteriormente, correspondientes a cada casete de expresión; a excepción de los cebadores para amplificar un fragmento de 114 pb de actina (5'- TCCTGATGGGCAAGTGATTAC-3' y 5'-TTGTATGTGGTCTCGTGGATTC-3'), diseñados por Liu et al., (2012) que fueron usados solamente en la PCR de RNA de las muestras CspB (2, 31, 3, 15, 40, 58 y 67) y DREB (9, 12, 25, 47 y 48) y para el control de la PCR de los cADNs. Por otra parte, teniendo en cuenta que el casete de expresión TaDREB3 fue diseñado con un promotor inducible por sequía, se obtuvo cADN de plantas TaDREB3 elongadas en medio MS y en medio MS + PEG (PM 6000) 10 %, que simula el estrés por sequía.
Evaluaciones fenotípicas in vitro
En el primer ensayo de transformación, la selección se llevó a cabo con 0,8 mg/l de PPT (Jimenéz, 2013). En el ensayo con CSPB se tomó la mayor cantidad de brotes PPT resistentes, varios por explante. En el ensayo con TaDREB3 y con AtZAT10 se tomó un único brote por explante, con el propósito de asegurar eventos de transformación independientes. Para corregir el efecto de escapes por baja presión de selección, se repitió el ensayo de transformación AtZAT10 incrementando la concentración del PPT a 2 mg/l.
Las pruebas fenotípicas para estrés por sequía in vitro se realizaron durante 20 días, con plántulas positivas para PCR y RT-PCR que expresan CspB, TaDREB3 y AtZAT10 cultivadas en medio MS + PEG10% (PM 6000), al mismo tiempo que plántulas control no transformadas (Shen et al., 2002). Estas plantas se mantuvieron en cuarto de crecimiento en condiciones de 70 % humedad relativa, 28 °C de temperatura y fotoperiodo de 16/8. Cuando hubo necesidad de realizar análisis estadístico se usó el Z-Test (p<0,05).
RESULTADOS
Diseño de genes semisintéticos
Los casetes de expresión CspB, TaDREB3 y AtZAT10, diseñados in silico corresponden a un total de 3,232 pb, 3,178 pb y 4,185 pb, respectivamente, incluyendo los sitios de restricción introducidos, la región espaciadora que separa los dos casetes de expresión, y ocho nucleótidos adicionales en ambos extremos de cada uno de los casetes de expresión para facilitar su inserción en el plásmido (Tab.1,2,3 y 4).
Usando la herramienta, marco abierto de lectura (ORF de sus siglas en inglés) del software Gen Designer 2.0, se identificó el ORF respectivo, se eligió la longitud nucleótidos correspondiente a la secuencia de la proteína obtenida en el NCBI o en patentes. A través de la herramienta "ORF finder" del NCBI se corroboró que el ORF de cada uno de los genes fuese el correcto. La comparación de la secuencia de aminoácidos que codifica la secuencia ORF diseñada, denominada como aminoácidos diseñados (AD), versus la secuencia de aminoácidos patrón, a través de un Clustal-W (EMBL-EBI, 2015) demostró un 100 % de identidad y similaridad y 0 % de gaps para el casete de expresión AtZAT10. Sin embargo, para CspB al inicio de la secuencia de la proteína presento una diferencia de ocho aminoácidos adicionales en comparación con la secuencia patrón, identificada en el Gen Bank como CAA42235.1, (con 67 aminoácidos), por lo tanto, se escindieron los primeros ocho codones de la secuencia de ADN, y de esta manera se logró la correspondencia de las secuencias aminoacídicas. De igual manera se escindió el primer codón para de la secuencia de ADN de TaDREB3, ya que se presentó un aminoácido adicional en comparación con la secuencia patrón. Con dichas modificaciones fue posible obtener un 100 % de identidad y similaridad y 0 % de gaps en todos los Clustal-W, adicionalmente se reconfirmó que el ORF no se cambiara después de las modificaciones.
La optimización de la región codificante de acuerdo al uso codónico de Z. mays, se realizó a través de la herramienta OPTIMIZER (OPTIMIZER, 2007), bajo la opción de criterio de optimización "DNA/ Guide Random" (Puigbo et al., 2007), teniendo en cuenta el G/C % y A/T %, índice de adaptación codónica (CAI) y número efectivo de codones (ENc). El CAI, mide el sesgo del uso codónico de un gen en relación a un conjunto de codones "óptimos" determinados a partir de un conjunto de referencia de genes de alta expresión, normalmente proteínas ribosomales (Sharp y Li, 1987). Los valores CAI se encuentran entre 0, cuando el uso codónico de una secuencia respecto al conjunto de referencia son muy diferentes, y uno, cuando ambos usos codónicos son iguales, mientras que los valores de ENc se encuentran en un rango de 20 (si solo un codón por aminoácido es usado) a 61 (si todos los codones sinónimos son usados igualmente) (Puigbo et al., 2007). Se les dio prioridad a los valores CAI, teniendo en cuenta que estos no fueran mayores a 0,8 y a los valores G/C % cercanos a 54,98, de acuerdo con la tabla de uso codónico de Kasusa, para Z. mays (Nakamura et al., 2000; Codon Usage Data Base, 2007). La secuencia optimizada del gen CspB de 204 nucleótidos; tiene 151 coincidencias, 36 transiciones (purina<_>purina/ primidina<_>pirimidina) y 17 transversiones (purinas<_>pirimidinas), respecto a la secuencia no optimizada (query). La secuencia optimizada del gen TaDREB3 de 675 nucleótidos, tiene 517 coincidencias, 62 transiciones y 96 transversiones, respecto a la secuencia no optimizada. La secuencia optimizada del gen AtZAT10 de 684 nucleótidos, tiene 497 coincidencias, 83 transiciones y 104 transversiones respecto a la secuencia no optimizada. Para todos los casetes de expresión se introdujo la misma secuencia del gen bar optimizada.
Tras la adición de sitios de restricción dentro del casete de expresión, que permiten aislar secuencias de interés del casete o hacer mapas de restricción; se realizó la reconfirmación del ORF del casete de expresión completo, que arrojó un marco de lectura correcto, tanto para el gen de tolerancia a sequía como para el gen de tolerancia a glufosinato de amonio. Finalmente la identidad de las secuencias nucleotídicas de los casetes de expresión completos fueron confirmadas a través Blastx (BLAST®), demostrándose 100 % de identidad en correspondencia a la descripción de la función de cada gen.
Caracterización fenotípica y molecular de las cepas recombinantes
Posterior a la transformación de las bacterias E. coli y A. tumefaciens, se realizó la siembra por agotamiento en el medio de selección. Las colonias que fueron sometidas al análisis de PCR, se subcultivaron en medio LB sólido, con el antibiótico de selección respectivo. El crecimiento de estas colonias en medio de selección, se considera la prueba fenotípica, puesto que no se evidenció crecimiento de bacterias no transformadas, cultivadas en el mismo medio de selección (control). Colonias de E. coli y de A. tumefaciens que fueron subcultivadas en medios de selección, resultaron positivas para PCR, amplificando los fragmentos predichos de los genes CspB (152 pb), TaDREB3 (181 pb) y AtZAT10 (212 pb), contenidos en los vectores de transformación, usados en los experimentos. Se observó que, para ambas cepas recombinantes, se presentó el mismo patrón de bandas en los análisis de electroforesis; las colonias transformadas con cada uno de los plásmidos también fueron PCR positivas para el gen bar (Fig. 2). Se consideran colonias positivas, cuando la banda correspondiente a los amplicones visualizada en la electroforesis en gel de agarosa y tinción con bromuro de etidio, corresponde al tamaño predicho de 352 pb para bar.
Transformación de plantas de N. benthamiana, caracterización molecular y fenotípica
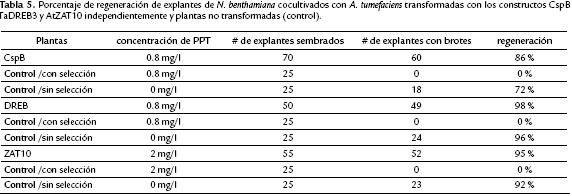
Se obtuvo un alto porcentaje de regeneración a partir de los explantes cocultivados y transferidos a medios de regeneración con selección por PPT, así como a partir de los explantes no cocultivados transferidos a medios de regeneración sin selección (control). De acuerdo con el resultado del Z-Test (p<0,05), se determinó que no hay diferencias significativas en el número de explantes que regeneraron plántulas, entre los dos grupos mencionados anteriormente, para cada uno de los casetes de expresión, correspondientes a ensayos independientes. Todos los explantes no cocultivados transferidos a medios de regeneración con selección, se tornaron blancos y no regeneraron plántulas.
Con el fin de determinar la presencia o la ausencia de los transgenes (CspB+bar, TaDREB3+bar y AtZAT10+bar), se realizó la caracterización molecular mediante PCR con el kit KAPA directamente sobre el tejido de hoja, con plántulas obtenidas a partir de explantes cocultivados con A. tumefaciens. En el primer ensayo de transformación con selección de 0,8 mg/l de PPT (Jimenéz, 2013), con CSPB se obtuvieron 52 % de plántulas PCR positivas, a partir de brotes cosechados que no fueron discriminados por explante. Con TaDREB3 y con AtZAT10 se obtuvieron 15 % y 0 % de plántulas PCR positivas respectivamente, seleccionando un solo brote por explante. En el segundo ensayo de transformación con AtZAT10 donde se incrementó la concentración de PPT a 2 mg/l (De Block et al., 1987), se logró incrementar el porcentaje de plántulas PCR positiva (Tab. 5).
De acuerdo con el resultado del Z-Test (p<0,05), hay diferencias significativas entre el número de plantas PCR positivas para DREB y ZAT10 provenientes de explantes seleccionados con 0,8mg/l y 2 mg/l de PPT, respectivamente. El fragmento esperado que resultó de la amplificación por PCR para CspB, TaDREB3 y AtZAT10, corresponde a 200 pb (5'-ATGCTCGAAGGGAAGGTC-3' y 5'-GCCTCCTTTGTCACATTAGCA-3', 459 pb (5'-GACAGCC GAACAAAAAGTCC-3' y 5'-AAAGTGCCACATCTCCTGCT-3') y 674 pb (5'-GGCGCTAGAGGCTCTGACTA-3' y 5'-GCAGTTTCA CTGGGAAGTCG-3), respectivamente. Se muestran los resultados para AtZAT10 (Fig. 3) resultados similares fueron obtenidos para los otros genes.
A partir de las plántulas PCR positivas, se tomaron al azar entre cinco a siete plantas para la extracción de ARN, tratado con DNAse I (Fig. 4), antes de ser sometido a PCR y utilizado posteriormente para la síntesis de cADN.
La amplificación del cADN se realizó con los primers correspondientes para CspB, TaDREB3, AtZAT10 y Bar (RT-PCR). Se muestran los resultados para TaDREB3, obtenidos a partir de plántulas crecidas en medio de cultivo con y sin PEG 10% (Fig. 5). La amplificación mediante RT-PCR correspondiente a los fragmentos de los genes CspB, AtZAT10 y Bar fue positiva.
Los ensayos de caracterización fenotípica bajo simulación del estrés por sequía (PEG 10 %) no muestran ventajas evidentes en las transformantes CspB, TaDREB3 o AtZAT10 (PCR y RT-PCR positivas) respecto al control, correspondiente a plantas no transformadas en medio de elongación con PEG 10 %(Fig. 6).
DISCUSIÓN
Genes de tolerancia a sequía y glufosinato de amonio en maíz
Los genes seleccionados para el diseño y la obtención de los casetes de expresión que fueron probados en la planta modelo N. Benthamiana, corresponden a genes ampliamente estudiados, que tiene soporte científico y que pueden llegar a conferir tolerancia a sequía en maíz. La participación de múltiples vías de estreses mediante la manipulación de la expresión de factores de transcripción específicos (FT) ha demostrado ser una estrategia promisoria en la mejora de la tolerancia a la sequía.
Los factores de transcripción de la familia DREB/CBF actúan en la modulación de un grupo de genes que definen las principales vías metabólicas de tolerancia a estreses (Long y Ort, 2010). DREB2 juega un papel importante en la expresión génica relacionada con la sequía, pero DREB2A de Arabidopsis induce débilmente genes corriente abajo cuando es sobre-expresado, y tiene un efecto mínimo en el incremento de la tolerancia al estrés, debido a posibles modificaciones post-traduccionales o a su rápida degradación por el proteosoma 26S (Qin et al., 2008). Con todo, la proteína DREB2A se hace constitutivamente activa (DREB2A-CA) a través de la delección específica de residuos, demostrando que la sobreexpresión de DREB2ACA aumenta significativamente la tolerancia a la sequía en Arabidopsis, al costo de retraso del crecimiento, como se ha evidenciado con la expresión constitutiva de otros DREBs (Sakuma et al., 2006a). Nakashima et al., (2014), demostraron que la sobre-expresión de DREB1A mejora tolerancia a frío y deshidratación, mientras que la sobreexpresión de DREB2ACA mejora la tolerancia a deshidratación, pero levemente al frío en plantas transgénicas de Arabidopsis. En contraste, la expresión ectópica constitutiva o inducible por estrés del ZmDREB2A de maíz (Qin et al., 2007) y de GmDREB2 de soya (Gycine max) (Chen et al., 2007) en Arabidopsis resulta en mejora en la tolerancia a sequía, calor y estrés salino de plantas transgénicas sin defectos en el crecimiento, se sugiere que ninguna requiere de modificaciones post-traduccionales (Yang et al., 2010). Morran et al., (2011), transformaron cebada (Hordeum vulgare) y trigo (Triticum aestivum) con TaDREB3 y TaDREB2 y evaluaron tolerancia a sequía y heladas con un promotor constitutivo (2X 35S) en cebada e inducible (pRab17) en trigo. La población transgénica de cebada con sobre-expresión constitutiva mostró crecimiento lento, retraso en la floración y menor rendimiento del grano en comparación con las no transgénicas. Sin embargo, ambas líneas transgénicas mostraron mayor supervivencia en condiciones severas de sequía frente al control. Por otra parte, las plantas de trigo transformadas con pRab17-TaDREB3 y pRab17-TaDREB2 no mostraron defectos en el fenotipo. Las primeras se recuperaron más rápidamente que las segundas después del periodo de sequía. Pese a que estructuralmente las proteínas TaDREB2 y TaDREB3 son muy diferentes, parecen regular genes similares de respuesta a sequia; el incremento de la supervivencia de las plantas transgénicas de trigo bajo sequia puede ser explicada por la regulación positiva de los genes que codifican para proteínas LEA, dehidrinas (DFN) y proteínas de respuesta a frio (COR) a niveles más altos de los que normalmente se expresan bajo este estrés (Morran et al., 2011). Por todo esto, TaDREB3 fue el gen seleccionado en este trabajo para el diseño de uno de los casetes de expresión.
Por otra parte, existe fuerte evidencia que la sobre-expresión de DREB activa genes adicionales no involucrados en tolerancia a sequía, que pueden producir efectos deletéreos en el fenotipo, tanto en el rendimiento, como en el crecimiento, en el número de fruto y semilla, y en el peso fresco de las plantas transgénicas bajo condiciones normales (Wang et al., 2003). Por ello, para el diseño del casete de expresión con el gen DREB, en este trabajo, se utilizó el promotor inducible por sequía rab17, que a diferencia del promotor inducible rd29A ha demostrado tener expresión basal en ausencia de estrés hídrico (patente US20120102592). Los resultados de RT-PCR con plantas transformadas con DREB, crecidas in vitro con y sin PEG 10 % confirmaron el papel y la especificidad de este promotor al inducirse por sequía, ya que la expresión a nivel de mARN de DREB solo se evidenció en las plantas sometidas al estrés. Se concluye que a pesar que las condiciones de cultivo in vitro involucran un estrés para la planta, no inducen la actividad del promotor rab17, a diferencia de lo informado para plantas de maíz (Z. mays) transformadas con AtCBF3 y el promotor inducible rd29A (Al-Abed et al., 2007).
Por otra parte, algunos estudios indican que la expresión constitutiva del regulador transcripcional ZAT10 no parece causar efectos negativos en el fenotipo de las plantas de N. bentamiana, mientras que otros si lo informan. Xiao et al., (2009), realizaron uno de los estudios más completos en arroz transgénico evaluado para resistencia a sequía en condiciones de campo, que involucró plantas transformadas con siete genes documentados en relación a la tolerancia a sequía (CBF3, SOS2, NCED2, NPK1, LOS5, ZAT10 y NHX1), cada uno bajo el control de promotor constitutivo Actin 1 y el homologo inducible por estrés de arroz HVA22. El rendimiento relativo y fertilidad relativa de la espiga fueron los criterios principales para evaluar el desempeño de la resistencia a sequía. En general, las familias transgénicas de los constructos HVA22P:CBF3, HVA22P:NPK1, Actin1:LOS5, HVA22P:LOS5, Actin1:ZAT10, and HVA22P:ZAT10 mostraron incremento en el rendimiento por planta y fertilidad de la espiga, significativamente mayor en comparación con la no transformadas; mientras que las familias transgénicas de los otros cuatro constructos (Actin1:SOS2, Actin1:NCED2, HVA22P:NCED2, and HVA22P:NHX1) no mostraron tolerancia a sequía de acuerdo con los criterios evaluados. Los genes LOS5 y ZAT10 mostraron relativamente superiores efectos que los otros cinco, en mejorar la resistencia a sequía de arroz transgénico bajo condiciones de campo. Este estudio no informa efectos negativos en el fenotipo, cuando ZAT10 es expresado constitutivamente. En el presente trabajo, las plantas de N. benthamiana transformadas con el constructo que contiene el gen AtZAT10 dirigido por el promotor constitutivo de maíz Ubi1 no mostraron ningún efecto negativo en el fenotipo. Una de las posibles explicaciones es que ZAT10 actúa en partes bajas de la vía de señalización de estrés, corriente abajo de DREB (Yang et al., 2010), al parecer regulando la expresión de genes involucrados en el sistema de defensa de ROS, como ascorbato peroxidasa 2 (APX2) y ferro-superoxide dismutase (FSD1) principalmente (Mittler et al., 2006). Sin embargo, estos resultados difieren de informes anteriores en los que se ha evidenciado que la expresión constitutiva de ZAT10 resulta en incremento en la tolerancia a sequía, pero afecta negativamente el crecimiento en plantas transformadas (Sakamoto et al., 2004; Mittler et al., 2006).
Las plantas de N. benthamiana transformadas con el casete de expresión CspB no mostraron efectos negativos en el fenotipo pese a su expresión constitutiva. El gen CspB es el responsable de la tolerancia a sequía en maíz, del primer cultivo GM liberado comercialmente, desarrollado por la compañía Monsanto, el híbrido transgénico Droughtgard ™ (evento MON 87460), que fue liberado en el año 2013 en Estados Unidos. El evento MON 87460 fue donado por Monsanto al proyecto público-privado WEMA, diseñado para entregar el primer cultivo transgénico tolerante a sequía en maíz en África para el 2017 (James, 2014).
El gen bar de Streptomyces hygroscopicus que confiere resistencia a glufosinato de amonio, usado en los tres casetes de expresión desarrollados en este trabajo, ha mostrado ser un sistema muy eficiente de selección en los protocolos de transformación genética (White et al., 1990). Tiene una amplia historia de uso como marcador de selección y en cultivos GM tolerantes a este herbicida, incluyendo maíz (Gordom-Kamm et al., 1990), arroz (Christou et al., 1991), trigo (Vasil et al., 1992), remolacha azucarera (Beta vulgaris) (D'Halluin et al., 1992), canola (Brassica napus) y alfalfa (Medicago sativa) (Cobb, 1992), algodón (Gossypium hirsutum) (Keller et al., 1997), lechuga (Lactuca sativa) (Mohapatra et al., 1999), caña de azúcar (Saccharum officinarum) (Falco et al., 2000), yuca (Manihot esculenta) (Sarria et al., 2000) y fríjol (Phaseolus vulgaris) (Aragão et al., 2002). En el mundo los cultivos comerciales aprobados, tolerantes a glufosinato incluyen remolacha azucarera, canola, soya, arroz y maíz. Unas pocas líneas híbridas de maíz resistentes están registradas por Dekalb (adquirida por Monsanto), Mycogen (adquirida por Dow Agrosciences) y Syngenta. Una variedad de achicoria (Cichorium intybus) con esterilidad masculina que lleva el gen se registró por Bejo Zaden, NL (PatentLens, 2007).
Transformación y caracterización molecular/ fenotípica de cepas bacterianas y plantas
Se realizaron ensayos de PCR de colonia, sobre cepas resistentes a antibióticos luego de experimentos de transformación, para determinar la presencia de los genes de interés y de los casetes de expresión. Estos ensayos se hicieron sobre E. coli y A. tumefaciens. El total de las cepas evaluadas por PCR correspondió a cepas recombinantes en las que parece mantenerse la integridad de los casetes de expresión.
Los ensayos de transformación de plantas de N. benthamiana permitieron el establecimiento de un protocolo eficiente, mediante el uso de cefotaxime en los lavados de los explantes cocultivados para eliminar el exceso de bacteria, y el uso de carbenicilina en el medio de regeneración. Fue evidente la reducción de la contaminación por reactivación de la bacteria, posiblemente debida a la exposición a los dos antibióticos (Horsch et al., 1985; Silva y Fukai, 2001). El incremento en la concentración de selección a 2 mg/l de PPT, no afecta la regeneración comparada con explantes seleccionados a 0,8 mg/l, y se obtuvo un incremento en la eficiencia de transformación, debido a la reducción de escapes.
Los genes fueron modificados favoreciendo el uso codónico del maíz. Se espera que la expresión en N. benthamiana sea menor y tenga efecto limitado en el fenotipo bajo condiciones de estrés por sequía. Mittler et al., (2006), determinaron que plantas transgénicas con expresión constitutiva de ZAT10 podrían ser más tolerantes a estrés osmótico. Análisis de RNA blot demostraron que ZAT10 es expresado en respuesta a estrés hídrico (Sakamoto et al., 2000); congruentemente un estudio con microarreglos reveló que ZAT10 es regulado positivamente por sequía (Huang et al., 2008). Genes de soya que codifican para los ortólogos de Arabidopsis DREB1A y DREB1D de la familia AP2_EREBO y ZAT10/STZ de la familia C2H2_Zn inducidos por estrés por sequía, parecen estar involucrados en la regulación de respuesta a sequía durante crecimiento vegetativo en lugar de crecimiento reproductivo (Le et al., 2012).
Diseño de genes
El diseño de genes in silico es una estrategia eficiente para desarrollar casetes de expresión funcionales (Villalobos et al., 2006), puede ser más económica comparada con la clonación de genes en el laboratorio, ya que requiere menos tiempo y reactivos, debido a que no se precisa aislar genes y regiones regulatorias, ni el uso de vectores intermediarios, ni de enzimas de restricción y de modificación. Actualmente, los costos en la contratación de síntesis de genes han disminuido notablemente, dependen de la longitud del casete de expresión y pueden incluir su introducción en el vector de transformación. Otra de las ventajas del diseño de genes in silico es la disponibilidad de herramientas en línea, incluso gratuitas, que permiten identificar el ORF del gen, optimizar su uso codónico, traducirlo y compararlo con otras secuencias de bases de datos de genes y proteínas, que minimiza las posibilidades de error en la transcripción y traducción in vivo.
CONCLUSIONES
El diseño in silico de genes es una herramienta que favorece el desarrollo de casetes de expresión ya que permite modificar y organizar secuencias in silico tomadas a partir de bases de datos. Una de los aspectos clave del diseño in silico de genes es la posibilidad de modificar el uso codónico para favorecer la expresión de genes foráneos en una especie determinada. Los resultados de este estudio indican que el diseño in silico de los casetes de expresión es eficiente ya que se identificaron plantas transgénicas mediante PCR y se demostró que los transgenes correspondientes a CspB, TaDREB3, AtZAT10 y bar se están expresando. La presencia de plántulas resistentes a PPT es una evidencia indirecta de la expresión del gen bar. Los ensayos fenotípicos no muestran resultados contundentes respecto a la tolerancia a sequía al comparar las plantas transformadas con las plantas control en medio de cultivo con PEG 10 %, sin embargo, se debe tener en cuenta que esto puede estar relacionado con que modificación del uso codónico fue diseñada para maíz, por lo que la expresión en N. benthamiana puede ser menor, reflejándose en el efecto limitado sobre el fenotipo bajo condiciones de estrés por sequía. Sin embargo, en este mismo ensayo se demostró la eficiencia del promotor inducible por estrés en el caso del constructo rab17:TaDREB3.
AGRADECIMIENTOS
Los autores agradecen a la Universidad Nacional de Colombia, a la Federación Nacional de Cultivadores de Cereales y Leguminosas (FENALCE) y a Colciencias por financiar este proyecto.
REFERENCIAS
Ashraf M. Inducing drought tolerance in plants: Recent advances. Biotechnol Adv. 2010;28:169-183. Doi: 10.1016/j.biotechadv.2009.11.005.
Al-Abed D, Madasamy P, Talla R, Goldman S, Rudrabhatia S. Genetic engineering of maize with the Arabidopsis DREB1A/CBF3 gene using split-seed explants. Crop Sciences. 2007;47(6):2390-2402. Doi: 10.2135/cropsci2006.11.0712.
Aragão FJ, Vianna GR, Albino M, Rech EL. Transgenic dry bean tolerant to the herbicide glufosinate ammonium. Crop Science. 2002;42(4):1298-1302. Doi: 10.2135/cropsci2002.1298.
BLAST. Blastx. U.S. The National Library of Medicine-National Center of Biotechnology Information 2014. Available at: http://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastx&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome.
Bolaños J, Edmeades GO. The importance of the anthesis-silking interval in breeding for drought tolerance in tropical maize. Field Crops Res. 1996;48(1):65-80. Doi:. doi:10.1016/0378-4290(96)00036-6.
Boyer JS, Westgate ME. Grain yields with limited water. J Exp Bot. 2004;55(407):2385-2394. Doi: 10.1093/jxb/erh219.
Cai Y, Hartnett B, Gustafsson C, Peccoud J. A syntactic model to design and verify synthetic genetic constructs derived from standard biological parts. Bioinformatics. 2007;23(20):2760-2767. Doi: 10.1093/bioinformatics/btm446.
Castiglioni P, Warner D, Bensen RJ, Anstrom DC, Harrison J, Stoecker M, Heard JE. Bacterial RNA chaperones confer abiotic stress tolerance in plants and improved grain yield in maize under water-limited conditions. Plant Phys. 2008;147(2):446-455. Doi:. 10.1104/pp.108.118828.
Ciftci-Yilmaz S, Mittler R. The zinc finger network of plants. Cell Mol Life Sci. 2008;65(7-8):150-1160. Doi: 10.1007/s00018-007-7473-4.
Cobb A. The transfer of herbicide resistance to crops. In: Herbicides and Plant Physiology. London, New York: Chapman and Hall;1992. p.145-151.
Codon Usage Data Base. Codon Usage Tabulated. NCBI-GenBank. 2007. Available at: http://www.kazusa.or.jp/codon/cgi-bin/showcodon.cgi?species=4577.
Change. Intergovermental Panel on Climate Change. Climate Change 2007: impacts, adaptation and vulnerability. New York: Cambridge University Press; 2007. p. 1-10.
Chen J-Q, Meng X-P, Zhang Y, Xia M, Wang X-P. Over-expression of OsDREB genes lead to enhanced drought tolerance in rice. Biotechnol Lett. 2008;30:2191-2198. Doi: 10.1007/s10529-008-9811-5.
Chen M, Wang QY, Cheng XG, Xu ZS, Li LC, Ye XG, Ma YZ. GmDREB2, a soybean DRE-binding transcription factor, conferred drought and high-salt tolerance in transgenic plants. Biochem Biophys Res Commun. 2007;353(2):299-305. Doi: 10.1016/j.bbrc.2006.12.027.
Christou P, Ford TL, Kofron M. Production of transgenic rice (Oryza sativa L.) plants from agronomically important indica and japonica varieties via electric discharge particle acceleration of exogenous DNA into immature zygotic embryos. Nat Biot. 1991;(9):957-962. Doi: 10.1038/nbt1091-957.
Davletova S, Rizhsky L, Liang H, Shengqiang Z, Oliver DJ, Coutu J, Mittler R. Cytosolic ascorbate peroxidase 1 is a central component of the reactive oxygen gene network of Arabidopsis. Plant Cell. 2005;17(1):268-281. Doi: https://doi.org/10.1105/ tpc.104.026971.
De Block M, Botterman J, Vandewiele M, Dockx J, Thoen C, Gossele V, Leemans J. Engineering herbicide resistance in plants by expression of a detoxifying enzyme. EMBO J. 1987;6(9): 2513-2518.
D'Halluin K, Bossut M, Bonne E, Mazur B, Leemans J, Botterman J. Transformation of sugarbeet (Beta vulgaris L.) and evaluation of herbicide resistance in transgenic plants. Nat Biot. 1992;10(3):309-314. Doi: 10.1038/nbt0392-309.
DNA 2.0. Inc. Gene Designer 2.0 Software. 2014. Available at: https://www.dna20.com/resources/genedesigner.
Edgerton MD. Increasing crop productivity to meet global needs for feed, food, and fuel. Plant Phys. 2009;149(1):7-13. Doi: https://doi.org/10.1104/pp.108.130195.
Edmeades GO. Progress in achieving and delivering drought tolerance in maize-An update. Ithaca, NY: ISAAA; 2013. p. 130.
EMBL-EBI. EMBOSS Needle. European Molecular Biology Laboratory. 2014. Available at: http://www.ebi.ac.uk/Tools/psa/emboss_needle/.
Englbrecht CC, Schoof H, Böhm S. Conservation, diversification and expansion of C2H2 zinc finger proteins in the Arabidopsis thaliana genome. BMC Genomics. 2004;5(1):39. Doi: 10.1186/1471-2164-5-39.
Falco MC, Neto AT, Ulian EC. Transformation and expression of a gene for herbicide resistance in a Brazilian sugarcane. Plant Cell Reports. 2000;19(12):1188-1194. Doi: 10.1007/s002990000253.
Gordon-Kamm WJ, Spencer TM, Mangano ML, Adams TR, Daines RJ, Start WG, Lemaux PG. Transformation of maize cells and regeneration of fertile transgenic plants. Plant Cell. 1990;2(7):603-618. Doi: https://doi.org/10.1105/tpc.2.7.603.
Gosal SS, Wani SH, Kang MS. Biotechnology and Drought tolerance. J Crop Improvement. 2009; 23:19-54. Doi: 10.1080/15427520802418251.
Grant RF, Jackson BS, Kiniry JR, Arkin GF. Water deficit timing effects on yield components in maize. Agronomy J. 1989;81(1):61-65. Doi: 10.2134/agronj1989.00021962008100010011x.
Horn G, Hofweber R, Kremer W, Kalbitzer HR. Structure and function of bacterial cold shock proteins. Cell Mol Life Sci. 2007;64(12):1457-1470. Doi: 10.1007/s00018-007-6388-4.
Horsch RB, Fry JE, Hoffmann NL, Eichholtz D, Rogers SA, Fraley RT. A simple and general method for transferring genes into plants. Science. 1985;227:1229-1231. Doi: 10.1126/science.227.4691.1229.
Hsieh TH, Lee JT, Charng YY, Chan MT. Tomato plants ectopically expressing Arabidopsis CBF1 show enhanced resistance to water deficit stress. Plant Phys. 2002;130(2):618-626. Doi: https://doi.org/10.1104/ pp. 006783.
Huang D, Wu W, Abrams SR, Cutler AJ. The relationship of droughtrelated gene expression in Arabidopsis thaliana to hormonal and environmental factors. J Exp Bot. 2008;59(11):2991-3007. Doi: 10.1093/jxb/ern155.
James C. Global Status of Commercialized Biotech/GM Crops: 2014. Ithaca: ISAAA; 2014. p. 32.
Jimenéz J. Diseño de genes semi-sintéticos que confieran tolerancia a herbicidas en soya. Tesis de maestría en ciencias-biología. Universidad Nacional de Colombia. 2014. p. 159.
Keller G, Spatola L, McCabe D. E. N. N. I. S, Martinell B, Swain W, John ME. Transgenic cotton resistant to herbicide bialaphos. Transgenic Res. 1997;6(6):385-392. Doi: 10.1023/A:1018483300902.
Lafitte H. Estreses abióticos que afectan el maíz. In: Paliwal L, Granados G, Laffite H, Violic A, Maratheé J, editors. El Maíz en los Trópicos: Mejoramiento y Producción. Roma: FAO; 2001. Available at: http://www.fao.org/docrep/003/X7650S.
Le DT, Nishiyama R, Watanabe Y, Tanaka M, Seki M, Yamaguchi-Shinozaki K, Tran LSP, et al. Differential gene expression in soybean leaf tissues at late developmental stages under drought stress revealed by genome-wide transcriptome analysis. PLoS One. 2012;7(11):e49522. Doi: 10.1371/journal.pone.0049522.
Lee H, Guo Y, Ohta M, Xiong L, Stevenson B, Zhu JK. LOS2, a genetic locus required for cold-responsive gene transcription encodes a bi‐functional enolase. EMBO J. 2002;21(11):2692-2702. Doi: 10.1093/emboj/21.11.2692.
Liu D, Shi L, Han C, Yu J, Li D, Zhang Y. Validation of reference genes for gene expression studies in virus-infected Nicotiana benthamiana using quantitative real-time PCR. PLoS One. 2012;7(9):e46451. Doi: 10.1371/journal.pone.0046451.
Liu Q, Kasuga M, Sakuma Y, Abe H, Miura S, Yamaguchi-Shinozaki K, Shinozaki K. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression, respectively, in Arabidopsis. Plant Cell. 1998;10(8):1391-1406. Doi: https://doi.org/10.1105/tpc.10.8.1391.
Long SP, Ort DR. More than taking the heat: crops and global change. Curr Opin Plant Biol. 2010;13(3):240-247. Doi: 10.1016/j.pbi.2010.04.008.
Lopato S, Bazanova N, Morran S, Milligan AS, Shirley N, Langridge P. Isolation of plant transcription factors using a modified yeast one-hybrid system. Plant Methods. 2006:2(1):3. Doi: 10.1186/1746-4811-2-3.
Meissner R, Michael AJ. Isolation and characterisation of a diverse family of Arabidopsis two and three-fingered C2H2 zinc finger protein genes and cDNAs. Plant Mol Biol. 1997;33(4):615-624. Doi: 10.1023/A:1005746803089.
Mittler R, Kim Y, Song L, Coutu J, Coutu A, Ciftci-Yilmaz S, Zhu JK. Gain-and loss-of-function mutations in Zat10 enhance the tolerance of plants to abiotic stress. FEBS letters. 2006;580(28):6537-6542. Doi: 10.1016/j.febslet.2006.11.002.
Mohapatra U, McCabe MS, Power JB, Schepers F, Van Der Arend A, Davey MR. Expression of the bar gene confers herbicide resistance in transgenic lettuce. Transgenic Res. 1999;8(1):33-44. Doi: 10.1023/A:1008891216134.
Morran S, Eini O, Pyvovarenko T, Parent B, Singh R, Ismagul A, Lopato S. Improvement of stress tolerance of wheat and barley by modulation of expression of DREB/CBF factors. Plant Biotech J. 2011;9(2):230-249. Doi: 10.1111/j.1467-7652.2010.00547.x.
Nakamura Y, Gojobori T, Ikemura T. Codon usage tabulated from international DNA sequence databases: status for the year 2000. Nucleic Acids Res. 2000;28(1):292-292. Doi: 10.1093/nar/28.1.292.
Nakashima K, Yamaguchi-Shinozaki K, Shinozaki K. The transcriptional regulatory network in the drought response and its crosstalk in abiotic stress responses including drought, cold, and heat. Front Plant Sci. 2014;5:170. Doi: 10.3389/fpls.2014.00170.
Ngugi K, Cheserek J, Muchira C, Chemining'wa G. Anthesis to Silking Interval Usefulness in Developing Drought Tolerant Maize. J Ren Agriculture. 2013:84:90. Doi: 10.12966/jra.08,03.2013.
Oh SJ, Kwon CW, Choi DW, Song SI, Kim JK. Expression of barley HvCBF4 enhances tolerance to abiotic stress in transgenic rice. Plant Biotechnol J. 2007;5(5):646-656. Doi: 10.1111/j.1467-7652.2007.00272.x.
Oh SJ, Song SI, Kim YS, Jang HJ, Kim SY, Kim M, Kim JK. Arabidopsis CBF3/DREB1A and ABF3 in transgenic rice increased tolerance to abiotic stress without stunting growth. Plant Physiology. 2005;138(1):341-351. Doi: https://doi.org/10.1104/pp.104.059147.
Ohta M, Matsui K, Hiratsu K, Shinshi H, Ohme-Takagi M. Repression domains of class II ERF transcriptional repressors share an essential motif for active repression. Plant Cell. 2001;3(8):1959-1968. Doi: 10.1105/TPC.010127.
OPTIMIZER. Web server for optimizing the codon usage of DNA sequences. 2007. Universitat Rovira i Virgili. Available at: http://genomes.urv.cat/OPTIMIZER/obtimized.php.
PatentLens. Chapter 2 Introduction: The bar gene de PatentLens. 2007. Available at: http://www.cambia.org/daisy/Phosph/g2/710.html.
Pellegrineschi A, Reynolds M, Pacheco M, Brito RM, Almeraya R, Yamaguchi- Shinozaki K, Hoisington D. Stress-induced expression in wheat of the Arabidopsis thaliana DREB1A gene delays water stress symptoms under greenhouse conditions. Genome. 2004;47(3):493-500. Doi: 10.1139/g03-140.
Puigbo P, Guzman E, Romeu A, Garcia-Vallve S. OPTIMIZER: a web server for optimizing the codon usage of DNA sequences. Nucleic Acids Res. 2007;35(suppl 2):W126-W131. Doi: 10.1093/nar/gkm219.
Qin F, Sakuma Y, Tran LSP, Maruyama K, Kidokoro S, Fujita Y, Yamaguchi-Shinozaki K. Arabidopsis DREB2A-interacting proteins function as RING E3 ligases and negatively regulate plant drought stress-responsive gene expression. Plant Cell. 2008;20:1693-1707. Doi: https://doi.org/10.1105/tpc.107.057380.
Qin F, Kakimoto M, Sakuma Y, Maruyama K, Osakabe Y, Tran LSP, Yamaguchi‐Shinozaki K. Regulation and functional analysis of ZmDREB2A in response to drought and heat stresses in Zea mays L. Plant J. 2007;50(1):54-69. Doi: 10.1111/j.1365-313X.2007.03034.x.
Rizhsky L, Davletova S, Liang H, Mittler R. The zinc finger protein Zat12 is required for cytosolic ascorbate peroxidase 1 expression during oxidative stress in Arabidopsis. J Biol Chem. 2004;279(12):11736-11743.
Rozen S, Skalestky H. Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S, editors. Bioinformatics Methods and Protocols: Methods Mol Biol. Totowa: Humana Press; 2000. p. 365-386.
Sakamoto H, Araki T, Meshi T, Iwabuchi, M. Expression of a subset of the Arabidopsis Cys 2/His 2-type zinc-finger protein gene family under water stress. Gene. 2000;248(1):23-32. Doi: 10.1016/S0378-1119(00)00133-5.
Sakamoto H, Maruyama K, Sakuma Y, Meshi T, Iwabuchi M, Shinozaki K, Yamaguchi-Shinozaki K. Arabidopsis Cys2/His2-type zinc-finger proteins function as transcription repressors under drought, cold, and high-salinity stress conditions. Plant Physiol. 2004;136(1):2734-2746. Doi: https://doi.org/10.1104/pp.104.046599.
Sakuma Y, Maruyama K, Osakabe Y, Qin F, Seki M, Shinozaki K, Yamaguchi- Shinozaki K. Functional analysis of an Arabidopsis transcription factor, DREB2A, involved in drought-responsive gene expression. Plant Cell. 2006a;18(5):1292-1309. Doi: 10.1105/tpc.105.035881.
Sakuma Y, Maruyama K, Qin F, Osakabe Y, Shinozaki K, Yamaguchi-Shinozaki K. Dual function of an Arabidopsis transcription factor DREB2A in water-stress responsive and heat-stress-responsive gene expression. Proc Natl Acad Sci USA. 2006b;103(49):18822-18827. Doi: 10.1073/pnas.0605639103.
Sarria R, Torres E, Angel F, Chavarriaga P, Roca WM. Transgenic plants of cassava (Manihot esculenta) with resistance to Basta obtained by Agrobacterium-mediated transformation. Plant Cell Rep. 2000;19(4):339-344. Doi: 10.1007/s002990050737.
Schachtman DP, Goodger J. Chemical root to shoot signaling under drought. Trends Plant Sci. 2008;13(6):281-287. Doi: 10.1016/j.tplants.2008.04.003.
Shao HB, Chu LY, Jaleel CA, Zhao CX. Water-deficit stress-induced anatomical changes in higher plants. C R Biol. 2008;331: 215-225. Doi: 10.1016/j.crvi.2008.01.002.
Sharp PM, Li WH. The codon adaptation index-a measure of directional synonymous codon usage bias, and its potential applications. Nucleic Acids Res. 1987;15(3):1281-1295. Doi: 10.1093/nar/15.3.1281.
Shen YG, Du BX, Zhang WK, Zhang JS, Chen S. Y. AhCMO, regulated by stresses in Atriplex hortensis, can improve drought tolerance in transgenic tobacco. Theor App Genet. 2002:105(6-7):815-821.
Silva JD, Fukai S. The impact of carbenicillin, cefotaxime and vancomycin on chrysanthemum and tobacco TCL morphogenesis and Agrobacterium growth. J Appl Hort. 2001;3(1):3-12.
Statista. Distribution of global corn production in 2013, by country. Statista. 2015. Available at: http://www.statista.com/statistics/254294/distribution-ofglobal-corn-production-by-country-2012/.
Taberlet P, Gielly L, Pautou G, Bouvet J. Universal primers for amplification of three non-coding regions of chloroplast DNA. Plant Mol Biol. 1991;17(5):1105-1109. Doi: 10.1007/BF00037152.
Vasil V, Castillo AM, Fromm ME, Vasil IK. Herbicide resistant fertile transgenic wheat plants obtained by microprojectile bombardment of regenerable embryogenic callus. Nat Biotechnol. 1992;10(6):667-674. Doi: 10.1038/nbt0692-667.
Villalobos A, Ness JE, Gustafsson C, Minshull J, Govindarajan S. Gene Designer: a synthetic biology tool for constructing artificial DNA segments. BMC bioinformatics. 2006;7(1):285. Doi: 10.1186/1471-2105-7-285.
Wang JW, Yang FP, Chen XQ, Liang RQ, Zang LQ, Geng DM, Zhang XD, Song YZ, Zhang GS. Induced expression of dreb transcriptional factor and study on its physiological effects of drought tolerance in transgenic wheat. Acta Genetica Sinica. 2006;33(5):468-476. Doi: 10.1016/S0379-4172(06)60074-7.
Wang W, Vinocur B, Altman A. Plant responses to drought, salinity and extreme temperatures: towards genetic engineering for stress tolerance. Planta. 2003;218(1):1-14. Doi: 10.1007/s00425-003-1105-5.
Willimsky G, Bang H, Fischer G, Marahiel MA. Characterization of cspB, a Bacillus subtilis inducible cold shock gene affecting cell viability at low temperatures. J Bacteriol. 1992;174(20):6326-6335.
White J, Chang SY, Bibb MJ, Bibb MJ. A cassette containing the bar gene of Streptomyces hygroscopicus: a selectable marker for plant transformation. Nucleic Acids Res. 1990;18(4):1062.
Xiao BZ, Chen X, Xiang CB, Tang N, Zhang QF, Xiong LZ. Evaluation of seven function-known candidate genes for their effects on improving drought resistance of transgenic rice under field conditions. Mol Plant. 2009;2(1):73-83. Doi: 10.1093/mp/ssn068.
XiaoYan H, Ming C, HuiJun X, ShiQing G, XianGuo C, LianCheng L, et al. Obtaining of transgenic wheats with GH-DREB gene and their physiological index analysis on drought tolerance. Southwest China Journal of Agricultural Sciences. 2005;18(5):616-620. Doi:20063004178.
Yamaguchi-Shinozaki K, Shinozaki K. A novel cis-acting element in an Arabidopsis gene is involved in responsiveness to drought, low-temperature, or high-salt stress. Plant Cell. 1994;6(2): 251-264. Doi: 10.1105/tpc.6.2.251.
Yang S, Vanderbeld B, Wan J, Huang Y. Narrowing down the targets: towards successful genetic engineering of drought-tolerant crops. Mol Plant. 2010;3(3):469-490. Doi: 10.1093/mp/ssq016.
Zhang S, Li N, Gao F, Yang A, Zhang J. Over-expression of TsCBF1 gene confers improved drought tolerance in transgenic maize. Mol Breed. 2010;26(3):455-465. Doi: 10.1007/s11032-009-9385-5.
Referencias
Ashraf M. Inducing drought tolerance in plants: Recent advances. Biotechnol Adv. 2010;28:169-183. Doi:10.1016/j.biotechadv.2009.11.005
Al-Abed D, Madasamy P, Talla R, Goldman S, Rudrabhatia S. Genetic engineering of maize with the Arabidopsis DREB1A/CBF3 gene using split-seed explants. Crop Sciences. 2007;47(6):2390-2402. Doi: 10.2135/cropsci2006.11.0712
Aragão FJ, Vianna GR, Albino M, Rech EL. Transgenic dry bean tolerant to the herbicide glufosinate ammonium. Crop Science. 2002;42(4):1298-1302. Doi:10.2135/cropsci2002.1298
BLAST. Blastx. U.S. The National Library of Medicine-National Center of Biotechnology Information 2014. Available at: http://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastx&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome
Bolaños J, Edmeades GO. The importance of the anthesis-silking interval in breeding for drought tolerance in tropical maize. Field Crops Res. 1996;48(1):65-80. Doi:doi:10.1016/0378-4290(96)00036-6
Boyer JS, Westgate ME. Grain yields with limited water. J Exp Bot. 2004;55(407):2385-2394. Doi:10.1093/jxb/erh219
Cai Y, Hartnett B, Gustafsson C, Peccoud J. A syntactic model to design and verify synthetic genetic constructs derived from standard biological parts. Bioinformatics. 2007;23(20):2760-2767. Doi:10.1093/bioinformatics/btm446
Castiglioni P, Warner D, Bensen RJ, Anstrom DC, Harrison J, Stoecker M, Heard JE. Bacterial RNA chaperones confer abiotic stress tolerance in plants and improved grain yield in maize under water-limited conditions. Plant Phys. 2008;147(2):446-455. Doi:10.1104/pp.108.118828
Ciftci-Yilmaz S, Mittler R. The zinc finger network of plants. Cell Mol Life Sci. 2008;65(7-8):150-1160. Doi:10.1007/s00018-007-7473-4
Cobb A. The transfer of herbicide resistance to crops. In: Herbicides and Plant Physiology. London, New York: Chapman and Hall;1992. p.145-151.
Codon Usage Data Base. Codon Usage Tabulated. NCBI-GenBank. 2007. Available at: http://www.kazusa.or.jp/codon/cgi-bin/showcodon.cgi?species=4577
Change. Intergovermental Panel on Climate Change. Climate Change 2007: impacts, adaptation and vulnaribility. ." Genebra, Suíça.; 2001. p.1-10.
Chen J-Q, Meng X-P, Zhang Y, Xia M, Wang X-P. Over-expression of OsDREB genes lead to enhanced drought tolerance in rice. Biotechnol Lett. 2008;30:2191-2198. Doi: 10.1007/s10529-008-9811-5
Chen M, Wang QY, Cheng XG, Xu ZS, Li LC, Ye XG, Ma YZ. GmDREB2, a soybean DRE-binding transcription factor, conferred drought and high-salt tolerance in transgenic plants. Biochem Biophys Res Commun. 2007;353(2):299-305. Doi:10.1016/j.bbrc.2006.12.027
Christou P, Ford TL, Kofron M. Production of transgenic rice (Oryza sativa L.) plants from agronomically important indica and japonica varieties via electric discharge particle acceleration of exogenous DNA into immature zygotic embryos. Nat Biot. 1991;(9):957-962. Doi: 10.1038/nbt1091-957
Davletova S, Rizhsky L, Liang H, Shengqiang Z, Oliver DJ, Coutu J, Mittler R. Cytosolic ascorbate peroxidase 1 is a central component of the reactive oxygen gene network of Arabidopsis. Plant Cell. 2005;17(1):268-281. Doi:http:/ / dx. doi. org/ 10. 1105/ tpc. 104. 026971
De Block M, Botterman J, Vandewiele M, Dockx J, Thoen C, Gossele V, Leemans J. Engineering herbicide resistance in plants by expression of a detoxifying enzyme. EMBO J. 1987;6(9): 2513-2518.
D'Halluin K, Bossut M, Bonne E, Mazur B, Leemans J, Botterman J. Transformation of sugarbeet (Beta vulgaris L.) and evaluation of herbicide resistance in transgenic plants. Nat Biot. 1992;10(3):309-314. Doi:10.1038/nbt0392-309
DNA 2.0. Inc. Gene Designer 2.0 Software. 2014. Available at: https://www.dna20.com/resources/genedesigner
Edgerton MD. Increasing crop productivity to meet global needs for feed, food, and fuel. Plant Phys. 2009;149(1):7-13. Doi:http://dx.doi.org/10.1104/pp.108.130195
Edmeades GO. Progress in achieving and delivering drought tolerance in maize–An update. Ithaca, NY: ISAAA; 2013. p. 130.
EMBL-EBI. EMBOSS Needle. European Molecular Biology Laboratory. 2014. Available at: http://www.ebi.ac.uk/Tools/psa/emboss_needle/
Englbrecht CC, Schoof H, Böhm S. Conservation, diversification and expansion of C2H2 zinc finger proteins in the Arabidopsis thaliana genome. BMC Genomics. 2004;5(1):39. Doi:10.1186/1471-2164-5-39
Falco MC, Neto AT, Ulian EC. Transformation and expression of a gene for herbicide resistance in a Brazilian sugarcane. Plant Cell Reports. 2000;19(12):1188-1194. Doi:10.1007/s002990000253
Gordon-Kamm WJ, Spencer TM, Mangano ML, Adams TR, Daines RJ, Start WG, Lemaux PG. Transformation of maize cells and regeneration of fertile transgenic plants. Plant Cell. 1990;2(7):603-618. Doi:http://dx.doi.org/10.1105/tpc.2.7.603
Gosal SS, Wani SH, Kang MS. Biotechnology and Drought tolerance. J Crop Improvement. 2009; 23:19-54. Doi:10.1080/15427520802418251
Grant RF, Jackson BS, Kiniry JR, Arkin GF. Water deficit timing effects on yield components in maize. Agronomy J. 1989;81(1):61-65. Doi:10.2134/agronj1989.00021962008100010011x
Horn G, Hofweber R, Kremer W, Kalbitzer HR. Structure and function of bacterial cold shock proteins. Cell Mol Life Sci. 2007;64(12):1457-1470. Doi:10.1007/s00018-007-6388-4
Horsch RB, Fry JE, Hoffmann NL, Eichholtz D, Rogers SA, Fraley RT. A simple and general method for transferring genes into plants. Science. 1985;227:1229-1231. Doi:10.1126/science.227.4691.1229
Hsieh TH, Lee JT, Charng YY, Chan MT. Tomato plants ectopically expressing Arabidopsis CBF1 show enhanced resistance to water deficit stress. Plant Phys. 2002;130(2):618-626. Doi:http://dx.doi. org/ 10. 1104/ pp. 006783
Huang D, Wu W, Abrams SR, Cutler AJ. The relationship of droughtrelated gene expression in Arabidopsis thaliana to hormonal and environmental factors. J Exp Bot. 2008;59(11):2991-3007. Doi:10.1093/jxb/ern155
James C. Global Status of Commercialized Biotech/GM Crops: 2014. ISAAA Brief No. 49. ISAAA. Ithaca, NY:ISBN 978-1-892456-59-1 editorial?: 2014. p.
Jimenéz J. Diseño de genes semi-sintéticos que confieran tolerancia a herbicidas en soya. Tesis de maestría en ciencias-biología. Universidad Nacional de Colombia. 2014. p.
Keller G, Spatola L, McCabe D. E. N. N. I. S, Martinell B, Swain W, John ME. Transgenic cotton resistant to herbicide bialaphos. Transgenic Res. 1997;6(6):385-392. Doi:10.1023/A:1018483300902
Lafitte H. Estreses abióticos que afectan el maíz. In: Paliwal L, Granados G, Laffite H, Violic A, Maratheé J, editors. El Maíz en los Trópicos: Mejoramiento y Producción. Roma: FAO; 2001. Available at: http://www.fao.org/docrep/003/X7650S
Le DT, Nishiyama R, Watanabe Y, Tanaka M, Seki M, Yamaguchi-Shinozaki K, Tran LSP, et al. Differential gene expression in soybean leaf tissues at late developmental stages under drought stress revealed by genome-wide transcriptome analysis. PLoS One. 2012;7(11):e49522. Doi: 10.1371/journal.pone.0049522
Lee H, Guo Y, Ohta M, Xiong L, Stevenson B, Zhu JK. LOS2, a genetic locus required for cold‐responsive gene transcription encodes a bi‐functional enolase. EMBO J. 2002;21(11):2692-2702. Doi:10.1093/emboj/21.11.2692
Liu D, Shi L, Han C, Yu J, Li D, Zhang Y. Validation of reference genes for gene expression studies in virus-infected Nicotiana benthamiana using quantitative real-time PCR. PLoS One. 2012;7(9):e46451. Doi:10.1371/journal.pone.0046451
Liu Q, Kasuga M, Sakuma Y, Abe H, Miura S, Yamaguchi-Shinozaki K, Shinozaki K. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression, respectively, in Arabidopsis. Plant Cell. 1998;10(8):1391-1406. Doi:http://dx.doi.org/10.1105/tpc.10.8.1391
Long SP, Ort DR. More than taking the heat: crops and global change. Curr Opin Plant Biol. 2010;13(3):240-247. Doi:10.1016/j.pbi.2010.04.008
Lopato S, Bazanova N, Morran S, Milligan AS, Shirley N, Langridge P. Isolation of plant transcription factors using a modified yeast one-hybrid system. Plant Methods. 2006:2(1):3. Doi:10.1186/1746-4811-2-3
Meissner R, Michael AJ. Isolation and characterisation of a diverse family of Arabidopsis two and three-fingered C2H2 zinc finger protein genes and cDNAs. Plant Mol Biol. 1997;33(4):615-624. Doi:10.1023/A:1005746803089
Mittler R, Kim Y, Song L, Coutu J, Coutu A, Ciftci-Yilmaz S, Zhu JK. Gain-and loss-of-function mutations in Zat10 enhance the tolerance of plants to abiotic stress. FEBS letters. 2006;580(28):6537-6542. Doi:10.1016/j.febslet.2006.11.002
Mohapatra U, McCabe MS, Power JB, Schepers F, Van Der Arend A, Davey MR. Expression of the bar gene confers herbicide resistance in transgenic lettuce. Transgenic Res. 1999;8(1):33-44. Doi:10.1023/A:1008891216134
Morran S, Eini O, Pyvovarenko T, Parent B, Singh R, Ismagul A, Lopato S. Improvement of stress tolerance of wheat and barley by modulation of expression of DREB/CBF factors. Plant Biotech J. 2011;9(2):230-249. Doi:10.1111/j.1467-7652.2010.00547.x
Nakamura Y, Gojobori T, Ikemura T. Codon usage tabulated from international DNA sequence databases: status for the year 2000. Nucleic Acids Res. 2000;28(1):292-292. Doi:10.1093/nar/28.1.292
Nakashima K, Yamaguchi-Shinozaki K, Shinozaki K. The transcriptional regulatory network in the drought response and its crosstalk in abiotic stress responses including drought, cold, and heat. Front Plant Sci. 2014;5:170. Doi:10.3389/fpls.2014.00170
Ngugi K, Cheserek J, Muchira C, Chemining’wa G. Anthesis to Silking Interval Usefulness in Developing Drought Tolerant Maize. J Ren Agriculture. 2013:84:90. Doi: 10.12966/jra.08,03.2013
Oh SJ, Kwon CW, Choi DW, Song SI, Kim JK. Expression of barley HvCBF4 enhances tolerance to abiotic stress in transgenic rice. Plant Biotechnol J. 2007;5(5):646-656. Doi:10.1111/j.1467-7652.2007.00272.x
Oh SJ, Song SI, Kim YS, Jang HJ, Kim SY, Kim M, Kim JK. Arabidopsis CBF3/DREB1A and ABF3 in transgenic rice increased tolerance to abiotic stress without stunting growth. Plant Physiology. 2005;138(1):341-351. Doi:http://dx.doi.org/10.1104/pp.104.059147
Ohta M, Matsui K, Hiratsu K, Shinshi H, Ohme-Takagi M. Repression domains of class II ERF transcriptional repressors share an essential motif for active repression. Plant Cell. 2001;3(8):1959-1968. Doi:10.1105/TPC.010127
OPTIMIZER. Web server for optimizing the codon usage of DNA sequences. 2007. Universitat Rovira i Virgili. Available at: http://genomes.urv.cat/OPTIMIZER/obtimized.php
PatentLens. Chapter 2 Introduction: The bar gene de PatentLens. 2007. Available at: http://www.cambia.org/daisy/Phosph/g2/710.html
Pellegrineschi A, Reynolds M, Pacheco M, Brito RM, Almeraya R, Yamaguchi- Shinozaki K, Hoisington D. Stress-induced expression in wheat of the Arabidopsis thaliana DREB1A gene delays water stress symptoms under greenhouse conditions. Genome. 2004;47(3):493-500. Doi:10.1139/g03-140
Puigbo P, Guzman E, Romeu A, Garcia-Vallve S. OPTIMIZER: a web server for optimizing the codon usage of DNA sequences. Nucleic Acids Res. 2007;35(suppl 2):W126-W131. Doi:10.1093/nar/gkm219
Qin F, Sakuma Y, Tran LSP, Maruyama K, Kidokoro S, Fujita Y, Yamaguchi-Shinozaki K. Arabidopsis DREB2A-interacting proteins function as RING E3 ligases and negatively regulate plant drought stress–responsive gene expression. Plant Cell. 2008;20:1693-1707. Doi:http://dx.doi.org/10.1105/tpc.107.057380
Qin F, Kakimoto M, Sakuma Y, Maruyama K, Osakabe Y, Tran LSP, Yamaguchi‐Shinozaki K. Regulation and functional analysis of ZmDREB2A in response to drought and heat stresses in Zea mays L. Plant J. 2007;50(1):54-69. Doi:10.1111/j.1365-313X.2007.03034.x
Rizhsky L, Davletova S, Liang H, Mittler R. The zinc finger protein Zat12 is required for cytosolic ascorbate peroxidase 1 expression during oxidative stress in Arabidopsis. J Biol Chem. 2004;279(12):11736-11743.
Rozen S, Skalestky H. Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S, editors. Bioinformatics Methods and Protocols: Methods Mol Biol. Totowa: Humana Press; 2000. p. 365-386.
Sakamoto H, Araki T, Meshi T, Iwabuchi, M. Expression of a subset of the Arabidopsis Cys 2/His 2-type zinc-finger protein gene family under water stress. Gene. 2000;248(1):23-32. Doi:10.1016/S0378-1119(00)00133-5
Sakamoto H, Maruyama K, Sakuma Y, Meshi T, Iwabuchi M, Shinozaki K, Yamaguchi-Shinozaki K. Arabidopsis Cys2/His2-type zinc-finger proteins function as transcription repressors under drought, cold, and high-salinity stress conditions. Plant Physiol. 2004;136(1):2734-2746. Doi:http://dx.doi.org/10.1104/pp.104.046599
Sakuma Y, Maruyama K, Osakabe Y, Qin F, Seki M, Shinozaki K, Yamaguchi- Shinozaki K. Functional analysis of an Arabidopsis transcription factor, DREB2A, involved in drought-responsive gene expression. Plant Cell. 2006a;18(5):1292-1309. Doi:10.1105/tpc.105.035881
Sakuma Y, Maruyama K, Qin F, Osakabe Y, Shinozaki K, Yamaguchi-Shinozaki K. Dual function of an Arabidopsis transcription factor DREB2A in water-stress responsive and heat-stress-responsive gene expression. Proc Natl Acad Sci USA. 2006b;103(49):18822-18827. Doi:10.1073/pnas.0605639103
Sarria R, Torres E, Angel F, Chavarriaga P, Roca WM. Transgenic plants of cassava (Manihot esculenta) with resistance to Basta obtained by Agrobacterium-mediated transformation. Plant Cell Rep. 2000;19(4):339-344. Doi:10.1007/s002990050737
Schachtman DP, Goodger J. Chemical root to shoot signaling under drought. Trends Plant Sci. 2008;13(6):281-287. Doi:10.1016/j.tplants.2008.04.003
Shao HB, Chu LY, Jaleel CA, Zhao CX. Water-deficit stress-induced anatomical changes in higher plants. C R Biol. 2008;331: 215-225. Doi:10.1016/j.crvi.2008.01.002
Sharp PM, Li WH. The codon adaptation index-a measure of directional synonymous codon usage bias, and its potential applications. Nucleic Acids Res. 1987;15(3):1281-1295. Doi:10.1093/nar/15.3.1281
ShenYG, Du BX, Zhang WK, Zhang JS, Chen S. Y. AhCMO, regulated by stresses in Atriplex hortensis, can improve drought tolerance in transgenic tobacco. Theor App Genet. 2002:105(6-7):815-821.
Silva JD, Fukai S. The impact of carbenicillin, cefotaxime and vancomycin on chrysanthemum and tobacco TCL morphogenesis and Agrobacterium growth. J Appl Hort. 2001;3(1):3-12.
Statista. Distribution of global corn production in 2013, by country. Statista. 2015. Available at: http://www.statista.com/statistics/254294/distribution-ofglobal-corn-production-by-country-2012/
Taberlet P, Gielly L, Pautou G, Bouvet J. Universal primers for amplification of three non-coding regions of chloroplast DNA. Plant Mol Biol. 1991;17(5):1105-1109. Doi:10.1007/BF00037152
Vasil V, Castillo AM, Fromm ME, Vasil IK. Herbicide resistant fertile transgenic wheat plants obtained by microprojectile bombardment of regenerable embryogenic callus. Nat Biotechnol. 1992;10(6):667-674. Doi:10.1038/nbt0692-667
Villalobos A, Ness JE, Gustafsson C, Minshull J, Govindarajan S. Gene Designer: a synthetic biology tool for constructing artificial DNA segments. BMC bioinformatics. 2006;7(1):285. Doi:10.1186/1471-2105-7-285
Wang JW, Yang FP, Chen XQ, Liang RQ, Zang LQ, Geng DM, Zhang XD, Song YZ, Zhang GS. Induced expression of dreb transcriptional factor and study on its physiological effects of drought tolerance in transgenic wheat. Acta Genetica Sinica. 2006;33(5):468-476. Doi:10.1016/S0379-4172(06)60074-7
Wang W, Vinocur B, Altman A. Plant responses to drought, salinity and extreme temperatures: towards genetic engineering for stress tolerance. Planta. 2003;218(1):1-14. Doi:10.1007/s00425-003-1105-5
Willimsky G, Bang H, Fischer G, Marahiel MA. Characterization of cspB, a Bacillus subtilis inducible cold shock gene affecting cell viability at low temperatures. J Bacteriol. 1992;174(20):6326-6335.
White J, Chang SY, Bibb MJ, Bibb MJ. A cassette containing the bar gene of Streptomyces hygroscopicus: a selectable marker for plant transformation. Nucleic Acids Res. 1990;18(4):1062
Xiao BZ, Chen X, Xiang CB, Tang N, Zhang QF, Xiong LZ. Evaluation of seven function-known candidate genes for their effects on improving drought resistance of transgenic rice under field conditions. Mol Plant. 2009;2(1):73-83. Doi:10.1093/mp/ssn068
XiaoYan H, Ming C, HuiJun X, ShiQing G, XianGuo C, LianCheng L, et al. Obtaining of transgenic wheats with GH-DREB gene and their physiological index analysis on drought tolerance. Southwest China Journal of Agricultural Sciences. 2005;18(5):616-620. Doi:20063004178
Yamaguchi-Shinozaki K, Shinozaki K. A novel cis-acting element in an Arabidopsis gene is involved in responsiveness to drought, low-temperature, or high-salt stress. Plant Cell. 1994;6(2): 251-264. Doi: 10.1105/tpc.6.2.251
Yang S, Vanderbeld B, Wan J, Huang Y. Narrowing down the targets: towards successful genetic engineering of drought-tolerant crops. Mol Plant. 2010;3(3):469-490. Doi:10.1093/mp/ssq016
Zhang S, Li N, Gao F, Yang A, Zhang J. Over-expression of TsCBF1 gene confers improved drought tolerance in transgenic maize. Mol Breed. 2010;26(3):455-465. Doi:10.1007/s11032-009-9385-5
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
CrossRef Cited-by
1. Julian Mora-Oberlaender, Jenny Jiménez-Barreto, Yadira Rodríguez-Abril, Meike Estrada-Arteaga, Alejandro Chaparro-Giraldo. (2022). Cisgenic Crops: Potential and Prospects. Concepts and Strategies in Plant Sciences. , p.89. https://doi.org/10.1007/978-3-031-06628-3_6.
Dimensions
PlumX
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2016 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).