Caracterización molecular de palma de aceite Elaeis guineensis Jacq., procedente de diferentes orígenes (Zaire y Camerún) usando marcadores microsatélites
Molecular characterization of oil palm Elaeis guineensis Jacq., from different origins (Zaire and Cameroon) using microsatellite markers
DOI:
https://doi.org/10.15446/acag.v65n3.48413Palabras clave:
Diversidad genética, estructura poblacional, conservación, heterocigocidad esperada y observada (es)Genetic diversity, population structure, conservation, the observed and expected heterozygosity. (en)
The genetic variability of 96 accessions of oil palm (E. guineensis Jacq.) from Cameroon and Zaire with 20 microsatellite markers was determined. 59 alleles were detected with an average of 2.95, three loci were polymorphic with a PIC (Polymorphic Information Index) average 0.58. High genetic diversity, with a total heterozygosity of 0.46, and a high percentage of polymorphic loci of 90% was found. FST value was of 0.14, suggesting that in the populations of Cameroon and Zaire there is a moderate population structure. The observed heterozygosity (Ho = 0.67) was higher than expected heterozygosity (He = 0.42), demonstrating a low presence of homozygous. The parameters of population differentiation as the FIS and FIT confirmed a high number of heterozygotes with respect to expected under the conditions of Hardy-Weinberg equilibrium. Microsatellite allowed discriminating genotypes according to the origin place. The results obtained show that the collections of Cameroon and Zaire may be considered as a single population, with moderate population structure despite of the constant existent gene flow. These results could be used in future conservation programs, selection and genetic improvement of the species.
Caracterización molecular de palma de aceite Elaeis guineensis Jacq., procedente de diferentes orígenes (Zaire y Camerún) usando marcadores microsatélites
Molecular characterization of oil palm Elaeis guineensis Jacq., from different origins (Zaire and Cameroon) using microsatellite markers
Carlos Felipe González Chavarro 1, Marlon Eduardo León Lozano 2, Ana Cruz Morillo Coronado 3, Erick Iván Ochoa 4 y Yacenia Morillo Coronado 2*
1Corporación Colombiana de Investigación Agropecuaria Corpoica, C.I. Tibaitatá, Mosquera, Cundinamarca. 2Universidad de los Llanos, Villavicencio, Meta, Colombia. 3Universidad Pedagógica y Tecnológica de Colombia, Tunja, Boyacá. 4Unipalma S.A, Villavicencio, Meta. *Autora para correspondencia: ymorillo@unillanos.edu.co
Rec.: 19.01.2015 Acep.: 30.07.2015
Resumen
Se determinó la variabilidad genética de 96 accesiones de palma de aceite (E. guineensis Jacq.) procedentes de Camerún y Zaire con 20 marcadores microsatélites. 59 alelos fueron detectados con un promedio de 2.95, tres loci fueron polimórficos con un PIC (Índice de Información Polimórfica) promedio de 0.58. Se encontró una alta diversidad genética, con una heterocigocidad total de 0.46, y un alto porcentaje de loci polimórficos del 90%. El valor de FST encontrado fue de 0.14, lo cual sugiere que en las poblaciones de Camerún y Zaire existe una moderada estructura poblacional. La heterocigocidad observada (Ho=0.67) fue mayor que la heterocigocidad esperada (He=0.42), evidenciando una baja presencia de homocigotos. Los parámetros de diferenciación poblacional como el FIS y el FIT confirmaron un alto número de heterocigotos con respecto a lo esperado bajo las condiciones de equilibrio Hardy–Weinberg. Los microsatélites permitieron discriminar los genotipos según el lugar de procedencia. Los resultados obtenidos ponen de manifiesto que las colecciones de Camerún y Zaire pueden ser consideradas como una sola población, con moderada estructuración poblacional a pesar del constante flujo génico existente. Estos resultados podrían ser utilizados en futuros programas de conservación, selección y mejoramiento genético de la especie.
Palabras clave: Diversidad genética, estructura poblacional, conservación, heterocigocidad esperada y observada.
Abstract
The genetic variability of 96 accessions of oil palm (E. guineensis Jacq.) from Cameroon and Zaire with 20 microsatellite markers was determined. 59 alleles were detected with an average of 2.95, three loci were polymorphic with a PIC (Polymorphic Information Index) average 0.58. High genetic diversity, with a total heterozygosity of 0.46, and a high percentage of polymorphic loci of 90% was found. FST value was of 0.14, suggesting that in the populations of Cameroon and Zaire there is a moderate population structure. The observed heterozygosity (Ho = 0.67) was higher than expected heterozygosity (He = 0.42), demonstrating a low presence of homozygous. The parameters of population differentiation as the FIS and FIT confirmed a high number of heterozygotes with respect to expected under the conditions of Hardy–Weinberg equilibrium. Microsatellite allowed discriminating genotypes according to the origin place. The results obtained show that the collections of Cameroon and Zaire may be considered as a single population, with moderate population structure despite of the constant existent gene flow. These results could be used in future conservation programs, selection and genetic improvement of the species.
Keywords: Genetic diversity, population structure, conservation, the observed and expected heterozygosity.
Introducción
La palma de aceite (Elaeis guineensis Jacq.), es una planta perenne tropical originaria en las Costas del Golfo de Guinea en el África Occidental y Central (Corley y Tinker, 2003). La palma de aceite es importante por la variedad de productos que genera, los cuales se utilizan en la alimentación y la industria. Tanto el aceite de pulpa como el de almendra se emplean para producir margarina, manteca, aceite de mesa y de cocina y jabones.
La utilidad de los marcadores moleculares en palma radica en la posibilidad de brindar apoyo a los programas de fitomejoramiento, mediante el reconocimiento y posterior selección de materiales élite en estado de pre vivero o vivero, acelerando así el mejoramiento convencional (Rocha, 2004). La variabilidad genética detectada por los marcadores moleculares a través de diferentes técnicas de biología molecular tiene su utilidad práctica al permitir la diferenciación entre un gran número de individuos al proporcionar información sobre las relaciones genéticas entre los diferentes organismos. Han demostrado ser una valiosa herramienta en la caracterización y evaluación de la diversidad genética entre y dentro de especies y poblaciones; poseen alto polimorfismo, frecuencia abundante en el genoma, comportamiento selectivamente neutral, fácil acceso y alta reproducibilidad No son afectados por el ambiente y no varían con la edad de la planta, además tienen la ventaja que la información genética es igual en todas las células somáticas.
Las secuencias sencillas repetidas ("SSR, Simple Sequence Repeats") posteriormente, denominadas microsatélites, constituyen la clase más polimórfica de marcadores moleculares hoy disponible. Son ampliamente utilizados debido a que presenFtan elevado contenido de información polimórfica, por su alta abundancia en el genoma, alta variabilidad debida a su elevado número de alelos por locus, por su forma de herencia codominante y no son afectados por el ambiente (Kalia et al., 2011).
Representan una herramienta poderosa para la identificación de genotipos élites que puedan ser incluidos en programas de mejoramiento (Hayati et al., 2004). Los microsatélites han sido utilizados ampliamente para la caracterización molecular de colecciones genéticas del género Elaeis (Arias et al., 2010; Billotte et al., 2001, 2005; Cochard et al., 2009; Singh et al., 2008).
El objetivo de este trabajo fue evaluar la diversidad genética de 96 accesiones de palma de aceite (Elaeis guineensis Jacq.) de las colecciones de Zaire y Camerún de Unipalma S.A., empleando 20 marcadores microsatélites.
Materiales y Métodos
Material Vegetal
Se evaluaron 96 accesiones de Palma, de las cuales 48 fueron de la Colección Zaire (actualmente conocido como Congo) y 48 de la Colección Camerún del Banco de Germoplasma de Unipalma S.A, localizado en la vereda Veracruz, Municipio de Cumaral (Departamento del Meta), situado a 4°13'33'' N; 73°14'50'' O , con una altitud de 452 m.s.n.m.
Análisis Molecular
De cada planta se obtuvo de tres a cuatro foliolos, en buen estado fitosanitario, se almacenaron en sílica gel y fueron transportadas al Laboratorio de Biología Molecular de la Universidad de los Llanos. Para la extracción de ADN se utilizó el protocolo de Dellaporta modificado previamente. El ADN genómico total se visualizó en geles de agarosa al 0.8% teñidos con Bromuro de etidio y visualizados con luz UV. La concentración de ADN de cada muestra se determinó por espectrofotómetro NanoDrop ND–1000. El ADN cuantificado se diluyó en agua destilada estéril en un volumen de 100 µl a 10 ng/µl y se almacenó a –20°C.
Se utilizaron 20 marcadores microsatélites los cuales han sido evaluados en estudios realizados por Billotte et al. (2001; 2005) y por Singh et al. (2008) y han sido útiles en la determinación de la diversidad genética en palma (Tabla 1).

La reacción de amplificación se preparó en un volumen final de 25 µl. La mezcla de reacción incluyó Buffer 1X, MgCl2 1.5 mM, dNTPs 0.2 mM, Taq Polimerasa 1U, cebador 2 µM y ADN genómico 10ng.
La amplificación se llevó a cabo en un termociclador (PxE 0,5 Thermal Cycler de Thermo Electron Corporation). La desnaturalización inicial fue de 94°C durante 2 minutos, seguidos por 30 ciclos de desnaturalización a 94 °C por 1 minuto, hibridación de 45 – 55 °C por un minuto (dependiendo del primer), una extensión a 72 °C por 2 minutos y la extensión final a 72 °C durante 5 minutos.
El producto amplificado se visualizó mediante electroforesis en gel de agarosa Metaphor al 4%, a 100 voltios durante una hora, y posteriormente se fotografió.
Análisis estadístico
La información de los patrones de bandas obtenidos se registró en una matriz numérica en donde se asignó un consecutivo para cada uno de los alelos encontrados por locus y a cada individuo se le asignó un máximo de dos valores por locus, dependiendo de su genotipo (homocigoto – heterocigoto). Para la selección de bandas polimórficas se consideró como locus polimórfico aquel en el cual la frecuencia del alelo más común fue menor al 95%. A partir de esta matriz y usando los programas NTSYS– PC (Numerical Taxonomy System for Personal Computer), TFPGA (Tools for Population Genetic Analysis) y Arlequin versión 3.11 se realizaron los análisis estadísticos.
El dendrograma fue construido a partir de la matriz de similaridad agrupando los datos con el método UPGMA (Unweighted Pair–Group Method Arithmetic Average), con el programa TREE de NTSYS–PC (versión 2.02®). Se calculó el coeficiente de correlación cofenética, que es una medida entre los valores de similitud del dendrograma y los de la matriz original de similaridad, mediante el programa COPH y MXCOMP del paquete NTSYS–PC. Se realizó un análisis de correspondencia múltiple (ACM) para asociar columnas y filas de la matriz binaria determinando el nivel de asociación o determinar proximidad.
Para estimar la diversidad genética se utilizaron los parámetros de heterocigocidad promedio esperada (He) y el porcentaje de loci polimórficos (P), los cuales se estimarán sobre todos los loci y el promedio de los mismos de acuerdo con la fórmula no sesgada de Nei:

Donde H: Probabilidad de que dos individuos tomados al azar tengan diferente alelo. H es el valor con que se representa la diversidad de la población.
f (i): Frecuencia del alelo i en la población.
Σ f(i) 2: Probabilidad de que dos individuos tomados al azar tengan el alelo i.
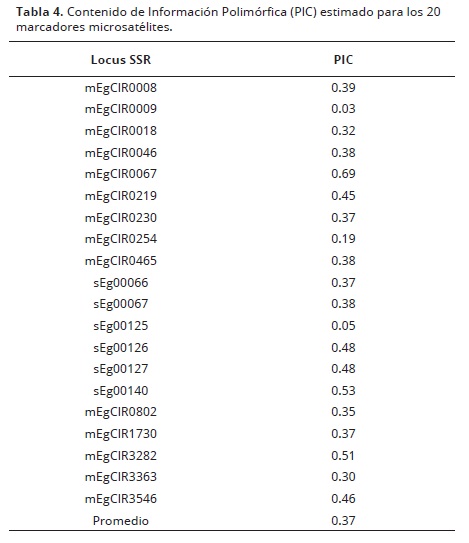
Finalmente, se calculó el contenido de información del polimorfismo (PIC) para los 20 loci analizados y se determinó posteriormente, la correspondiente estructura poblacional.
Resultados y Discusión
Caracterización molecular
Se obtuvo un total de 54 alelos, que fluctuaron entre 2 a 4 por locus y un promedio de 2.70 alelos/locus. Resultados similares fueron obtenidos por Singh et al. (2008) con microsatélites en el género Elaeis con un promedio de alelos de 2.56; Zaki et al. (2010), con 14 marcadores microsatélites en cuatro poblaciones de E. oleífera de diferentes países obtuvieron un valor de 2.66.
El porcentaje de loci polimórficos para Camerún fue del 100% frente a un 95% de Zaire. Con relación a las frecuencias alélicas, en un total de 59 alelos se obtuvieron valores de 0.05 (mEgCIR0018 y mEgCIR0465) a 0.98 (mEgCIR009 y mEgCIR0018) (Tabla 2). Para el alelo más frecuente Fr (A2) de cada marcador fue observada una variación de 0.010 (mEgCIR0018) a 0.875 (mEgCIR0254) y menos frecuente Fr (A5) de cada marcador tuvo una variación de 0.2343 (mEgCIR0067) a 0.6615 (sEg00127).
Cuando un alelo alcanza la frecuencia 1, se dice que se fija a la población y cuando alcanza la frecuencia 0 se ha perdido. Las frecuencias alélicas no varían más allá de los límites de 0 y 1, tarde o temprano, todas las familias deberán llegar a esos límites. Dado que la frecuencia alélica llega a 0 o 1 no puede cambiar su linaje, a no ser de sufrir mutación o migración.

Diversidad genética
La heterocigocidad observada (Ho) de las poblaciones evaluadas presentó valores entre 0.0 (mEgCIR0009) y 1.0 (mEgCIR0008, mEgCIR0018, mEgCIR0046, mEgCIR0219, mEgCIR0465, sEg00067 y sEg000126) tanto para Zaire como para Camerún, con un promedio de 0,67 (Tabla 3). La heterocigocidad esperada (He), que es la probabilidad de que un individuo sea heterocigoto para un loci, fue menor que la observada. Por tanto, se evidenció la baja presencia de homocigotos. En general, los valores de Ho fueron mayores que los de He en los loci evaluados donde los marcadores mEgCIR0008, mEgCIR0018, mEgCIR0046, mEgCIR0219, mEgCIR0465, sEg00067 y sEg000126, fueron los que presentaron elevada cantidad de heterocigotos.
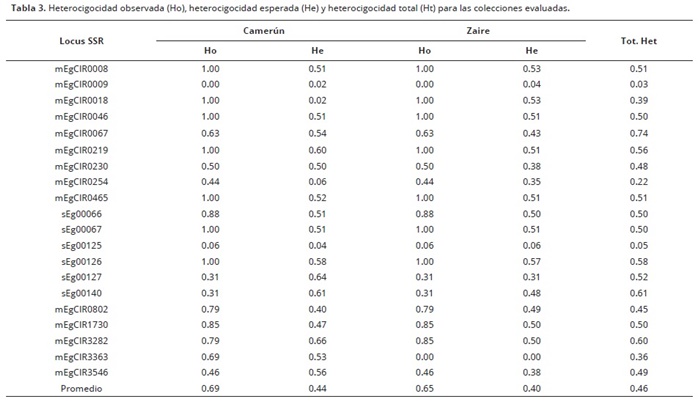
La heterocigocidad puede ser considerada como un indicativo de la existencia de variabilidad genética para especies alógamas como la palma. Los altos porcentajes de loci polimórficos, con una media de 90%, pueden ser atribuidos a la baja homocigocidad encontrada en diferentes genotipos.
La diversidad genética total (Ht) alcanzó una media de 0,46, lo que indica una alta diversidad genética del material evaluado similar a lo observado por (Allou et al., 2008), con cuatro loci microsatélites.
Contenido de Información Polimórfica (PIC)
El contenido de información polimórfica (PIC) se refiere a una medida del grado de información que brinda un determinado marcador genético, la cual depende del número de alelos para ese locus y de sus frecuencias relativas. Según Ott (1992), un locus es polimórfico cuando Ho fue superior a 0.1 y altamente polimórfico si Ho fue superior a 0.7.
De los 20 marcadores evaluados, los marcadores mEgCIR0009 y sEg00125 no fueron considerados polimórficos, 17 fueron considerados polimórficos y el marcador mEgCIR0067 resultó altamente polimórfico, presentando cinco alelos (Tabla 4).
Singh et al. (2008), utilizando diez marcadores microsatélites en palma de aceite, obtuvo valores de PIC entre 0,19 y 0,84. Así, el número de loci polimórficos es directamente proporcional al grado de heterocigocidad.
Diferenciación de poblaciones
El valor de FST detectado fue de 0,11 (Tabla 5), lo cual indica que existe una moderada diferenciación genética de las colecciones. El grado de diferenciación indica que el 11% de la variación se debe a diferencias entre poblaciones.
Montoya et al. (2005), reportaron un FST de 0,084 entre genotipos procedentes de Angola. Hayati et al. (2004) reportaron un FST de 0,301 entre las poblaciones de la Costa Oeste y Centro de África. En tanto que para la zona que abarcaba los países de República Democrática del Congo, Tanzania y Angola reportaron un FST= 0,073 (diferenciación genética moderada). Maizura et al. (2001), reportaron un FST= 0,175 en amplio rango de poblaciones africanas.
Las poblaciones naturales de organismos muestran casi siempre diferencias de frecuencias génicas y genotípicas en el espacio geográfico. Si esas diferencias llegan al extremo de ser significativas y las subpoblaciones no intercambian genes (en forma de migrantes o granos de polen que fecunden a individuos de otras subpoblaciones), entonces cada una de ellas puede evolucionar independientemente y con el tiempo dar lugar a nuevas especies. Evaluar la existencia de estructura genética poblacional es importante debido a que las poblaciones que presenten estructuración, pueden diversificar y diferenciarse genéticamente.
En el caso de las colecciones en la zona geográfica de procedencia, se encontró una moderada diferenciación genética, lo cual indica que posiblemente existe un grado de flujo génico entre las colecciones y por lo tanto, no existirían barreras ni marcada diferencia entre las colecciones. Lo anterior posiblemente se deba al transporte de materiales asociados con actividades humanas, polinización cruzada natural, o por eventos de migración de cepas o semillas por corrientes de agua, aves y otros animales.
El valor promedio del índice FIS obtenido fue de –0,53; comprendido en un rango de –0,01732 (sEg00125) y –1,0 (mEgCIR0046 y sEg00067), donde 18 marcadores de los 20 evaluados fueron negativos (Tabla 5). Este valor negativo confirmó un alto número de heterocigotos con respecto a lo esperado bajo las condiciones de equilibrio Hardy–Weinberg. Los 20 marcadores microsatélites se encontraron en equilibrio HW, a excepción del microsatélite mEgCIR0009, esto significa que la composición genética de una población natural permanece en equilibrio mientras no actúen la selección natural, la mutación o la deriva genética.
El exceso de heterocigotos encontrado en las dos colecciones puede ser explicado por una inmigración importante conducente a la heterogamia, selección a favor de individuos heterocigotos (selección sobredominante), la naturaleza alógama de la especie, el estado silvestre de los genotipos analizados, entre otros factores. Lo anterior se ve reflejado en el elevado valor de heterocigocidad encontrado en la población total (0.5), así como el grado de loci polimórficos (90%), lo cual es de suma importancia para explotar el potencial genético de la especie, mejorar la base genética del cultivo, diseñar germoplasma y la identificación de genotipos élites, los cuales podrían ser utilizados como parentales en el mejoramiento genético de la palma.
El valor de FIT fue de –0,35, lo cual refuerza el valor de FIS, por lo tanto, se detectó un marcado aumento del número de heterocigotos respecto a lo esperado en equilibrio de Hardy–Weinberg.
Dendrograma
A un nivel de similaridad del 80%, las colecciones de Camerún y Zaire, se diferenciaron en base a su procedencia geográfica (Figura 1). Se detectó una correlación cofenética elevada (r = 1.00), lo cual indica que el dendrograma representa fielmente los valores de la matriz de similitud. A un índice de similitud entre 92 y 98%, existe una alta concentración de genotipos, sugiriendo una alta similitud entre ellos, esto posiblemente se deba al constante flujo génico entre las colecciones en estudio.

El genotipo 5404 de Camerún presentó el menor valor de similaridad (86,5%) con respecto a los demás genotipos evaluados, posiblemente por características propias de esta especie tales como: tipo de material, origen de la progenie y tal vez características morfológicas.
Cochard et al. (2009), en un estudio de diversidad genética con 17 marcadores SSR, observaron una clara separación del grupo con origen Deli de aquellos procedentes de Costa de Marfil, como lo es el material La Mé y el de materiales provenientes de África Central.
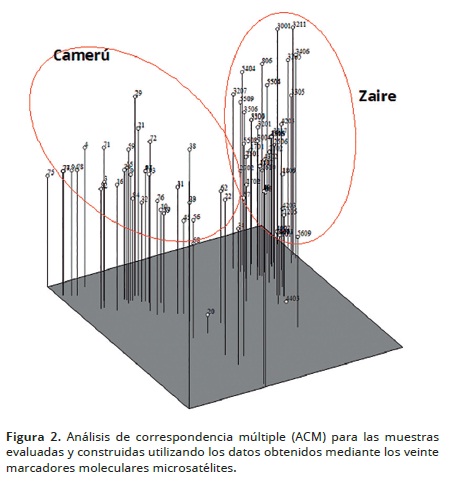
Análisis de correspondencia múltiple (ACM)
Los resultados confirman que existe una alta relación entre las zonas geográficas de procedencia de los materiales, además, muestra una alta variación de los materiales en las colecciones evaluadas y con identidad genética por lo cual forman grupos homogéneos (Figura 2). Se observa una clara distribución de las plantas entre las colecciones, agrupándose de acuerdo a su procedencia.
Conclusión
- La elevada variabilidad genética obtenida en los materiales de palma silvestres evaluados, puede ser utilizada en procesos de selección de parentales y recombinación genética con fines de obtener genotipos élites con características agronómicas deseadas, adaptación a diferentes zonas agroecológicas, incrementos en la producción de aceite, alto rendimiento y resistencia a enfermedades y plagas limitantes del cultivo.
Agradecimientos
A la Empresa Unipalma S.A., al Doctor Salvador Rojas González, el Laboratorio de Biología Molecular de Medicina Veterinaria y Zootecnia (MVZ) de la Universidad de los Llanos. Este trabajo de investigación fue seleccionado en la "Convocatoria para apoyar financieramente la ejecución de Proyectos de Investigación de grado año 2012", por la Dirección General de Investigaciones de la Universidad de los Llanos.
Referencias
Allou. D. Adon. B. & Sangare. A. (2008). Molecular Variability from two selection of BRT10 population in an inbreeding program of oil palm (Elaeis guineensis Jacq) in Cote d'Ivoire. Afr J Biotechnol, 7(20), 3550–3553. doi: 10.5897/AJB08.080.
Arias. D. M. Montoya. C. Romero. H. M. (2010). Preliminary results on the molecular characterization of oil palm using microsatellites markers. Palmas, 31(3), 35–45.
Billotte. N. Rusterucci. A. Barcelós. Noyer. J. Amblard. P. & Baurens. F. (2001). Development, characterization and across–taxa utility of oil palm (Elaeis guineensis Jacq) microsatellite markers. Genome, 44(3), 413–425. doi: 10.1139/g01–017.
Billotte. N. N. Marseillac. A. M. Risterucci. B. Adon. P. Brottier. F. C. Baurens. R. Charrier. A. (2005). Microsatellite–based high density linkage map in oil palm (E. guineensis Jacq). Theor Appl Genet, 110(4), 754–765. doi:10.1007/s00122–004–1901–8.
Cochard. B. Adon. B. Rekima. S. Billotte. N. Desmier. de Chenon. R. Koutou. A. Nouy. B. Omoré. A. Purba. A. R. Glazsmann. J. C. Noyer. J. L. (2009). Geographic and genetic structure of African oil palm diversity suggest new approaches to breeding. Tree Genet Genomes, 5(3), 493–504. doi: 10.1007/s11295–009–0203–3.
Cole. D. White. T. Nair. P. K. R. (2007). Maintaining genetic resources of peach palm (Bactris gasipaes Kunth): The role of seed migration and swidden–fallow management in northeastern Peru. Genet Resour Crop Ev, 54(1), 189–204. doi: 10.1007/s10722–005–3134–3.
Corley. R. & Tinker. P. (2003). La palma de aceite. 4th edición. (Fedepalma, Traducción.) Blackwell Science, Oxford, UK.
Kalia. R. K. Rai. M. K. Kalia. S. Singh. R. & Dhawan. A. K. (2011). Microsatellite markers: an overview of the recent progress in plants. Euphytica, 177(3), 309–334. doi: 10.1007/s10681–010–0286–9.
Mayes. S. Jack. P. L. Marshall. D. F. & Corley. R. H. V. (1997). Construction of a RFLP genetic linkage map for oil palm (Elaeis guineensis Jacq.). Genome, 40(1), 116–122. doi: 10.1139/g97–016.
Mohammadi. S. & Prasanna. B. (2003). Analysis of genetic diversity in crop plants–salient statistical tools and consederations. Crop Sci, 43(4), 1235–1248. doi: 10.2135/cropsci2003.1235.
Montoya. C. Arias. D. Rey. L. & Rocha. P. (2005). Diversidad genética de materiales Elaeis guineesis Jacq., procedentes de Angola. Fitot Col, 5(2):1–10.
Ochoa. E.I. Villegas. V.E. & Beebe. S.E. (1997). Identificación de marcadores moleculares RAPD asociados con la resistencia al Complejo Pudrición de Cogollo en palma de aceite (Elaeis guineensis Jacq.).Palmas, 8(2), 33–38.
Ott. J. (1992). Strategies for chacaracterizing highly polymorphic markers in human gene mapping. Am J Hum Genet, 51(2), 283–290.
Peakall. R. & Smouse. P. (2012). GenAlex 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update. Bioinformatics, 28(19), 2537–2539. doi: 10.1093/bioinformatics/bts460.
Piñero. D. Barahona. A. Eguiarte. L. Rocha–Olivares. A. & Salas–Linaza. R. (2008). La variabilidad genética de las especies: aspectos conceptuales y sus aplicaciones y perspectivas en México. Capital natural de México, I: Conocimiento actual de la biodiversidad (CONABIO). 415–435 p.
Purba. A. R. Noyer. J. L. Baudouin. L. Perrier. X. Hamon. S. Lagoda. P. J. L. (2000). A new aspect of genetic diversity of Indonesian oil palm (Elaeis guineensis Jacq.) revealed by isoenzyme and AFLP markers and its consequences for breeding. Theor Appl Genet, 101(5–6), 956–961. doi: 10.1007/s001220051567.
Rocha. P.J. (2004). Conceptos básicos en biotecnología de la palma de aceite. Palmas, 25(sup.esp), 11–17.
Shah. F. H. Rashid. O. Simons. A. J. Dundson. A. (1994). The utility of RAPD markers for determination of genetic variation in oil palm (Elaeis guineensis). Theor Appl Genet, 89(6), 713–718. doi: 10.1007/BF00223710.
Singh. R. Noorhariza. M. Z. Ting. N. C. Rozana. R. Tan. S. G. & Low. E. T. L. (2008). Exploiting an oil palm EST database for the development of gene–derived SSR markers and their exploitation for assessment of genetic diversity. Biologia, 63(2), 227–235. doi: 10.2478/s11756–008–0041–z.
Zaki. N. M. Ismail. I. Rosli. R. Chin. T. N. & Singh. R. (2010). Development and characterization of Elaeis oleifera microsatellite markers. Sains Malays, 39(6), 909–912.
Referencias
Allou, D.; Adon, B. y Sangare, A. 2008. Molecular Variability from two selection of BRT10 population in an inbreeding program of oil palm (Elaeis guineensis Jacq) in Cote d´Ivoire. African Journal of Biotechnology. 7(20):3550-3553.
Araya, E.; Alvarado, A. y Escobar, R. 2009. Use of DNA markers for fingerprinting compact clones and determining the genetic relationship between Elais oleífera germplasm origins. San Jose, (ASD, Ed.) Costa Rica: Agricultural Services & Development. 21 p.
Arias, D. M.; Montoya, C.; Romero, H. M. 2010. Preliminary results on the molecular characterization of oil palm using microsatellites markers. PALMAS 31(3):35–45.
Barcelós, E. 1998. Etude de la diversité génétique du genre elaeis (E. oleifera Kunth) Cortés et E. guineensis Jacq.) par marqueurs moléculaires (RFLP et AFLP). Tesis doctoral. Ecole Nationale Supérieure Agronomique de Montpellier, 137 p.
Billotte, N.; Rusterucci, A., Barcelós; Noyer, J.; Amblard, P. y Baurens, F. 2001. Development, characterization and across-taxa utility of oil palm (Elaeis guineensis Jacq) microsatellite markers. Genome. 44: 413-425.
Billotte, N.; N. Marseillac, A. M.; Risterucci, B.; Adon, P.; Brottier, F. C.; Baurens, R.; Charrier, A. 2005. Microsatellite-based high density linkage map in oil palm (E. guineensis Jacq). Theoretical and Applied Genetics. 110(4):754-765.
Botstein, D.; White, R. L.; Skolnick, M. y Davis, R. W. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. Am J. Human Genet. 32:314-331.
Caujapé-Castells, J. 2006. Brújula para botánicos desorientados en la genética de poblaciones. EXEGEN Ediciones. Las Palmas de Gran Canaria. España. 132 p.
Cochard, B.; Adon, B.; Rekima, S.; Billotte, N.; Desmier de Chenon, R.; Koutou, A.; Nouy, B.; Omoré, A. Purba, A. R.; Glazsmann, J. C.; Noyer, J. L. 2009. Geographic and genetic structure of African oil palm diversity suggest new approaches to breeding. Tree genetics and genomes. 5(3):493-504.
Cole, D.; White, T.; Nair, P. K. R. 2007. Maintaining genetic resources of peach palm (Bactris gasipaes Kunth): The role of seed migration and swidden-fallow management in northeastern Peru. Genetic Resources and Crop Evolution. 54:189-204.
Corley, R. y Tinker, P. 2003. La palma de aceite. 4th. (Fedepalma, Traducción.) Blackwell Science, Oxford, UK.
Crisci, J. y López, M. 1983. Introducción a la teoría y práctica de la taxonomía numérica. Serie de biología No. 26, Secretaría General de la Organización de los Estados Americanos (OEA). Programa Regional de Desarrollo Científico y Tecnológico Washington, D.C.: 132 p.
Chua, K. L.; Singh, R.; Cheah, S. C. 2001. Construction of oil palm (Elaeis guineensis Jacq.) linkage maps using AFLP markers. 2001 PIPOC International Palm Oil Congress–Cutting-edge technologies for sustained competitiveness. Proceeding (Agriculture). (Malasia). 461 – 467 p.
Dice, L. 1945. Measures of the amount of ecological association between species. Ecology 26:297-302.
Fedepalma. 2007. Aceite de palma en la salud humana. Editorial Kimpres Ltda. Bogotá. Colombia. 122 p.
Frankel, O.; Brown, A. y Burdon, J. 1996. The conservation of plant biodiversity. Cambridge: Cambridge University Press.
Jack, P.L.; Mayes, S. 1999. Use of molecular markers for oil palm breeding II. Use of DNA markers (RFLPs). Oleagineux. Francia, 48:1-8.
Joseph, J. D.; Anderson, R.; Tatham, R.; Black, W. 1992. Multivariate data Analysys with Readings. 3er ed. USA. 554 p.
Kalia, R. K.; Rai, M. K.; Kalia, S.; Singh, R. & Dhawan, A. K. 2011. Microsatellite markers: an overview of the recent progress in plants. Euphytica 177:309–334.
Li, C. y Horvitz, D. 1953. Some methods of estimating the inbreeding coefficient. Ame. J. Human. Genet. 5:107-117.
Litt, M. y Luty, J. 1989. A hypervariable microsatellite revealed by in vitro amplification of a dinucleotide repeat within the cardiac muscle actin gene. American Journal of Human Genetics. 44:397-401.
Mayes, S.; Jack, P. L.; Marshall, D. F. y Corley, R. H. V. 1997. Construction of a RFLP genetic linkage map for oil palm (Elaeis guineensis Jacq.). Genome. 40(1):116-122.
Mohammadi, S. and Prasanna, B. 2003. Analysis of genetic diversity in crop plants-salient statistical tools and consederations. Crop Sci. 43(4):1235-1248.
Montoya, C.; Arias, D.; Rey, L. y Rocha, P. 2005. Diversidad genética de materiales Elaeis guineesis Jacq procedentes de Angola. Fitotecnica Colombiana. (Colombia), 5(2):1-10.
Muñoz, J. E. 2011. Diversidad genética, estructura poblacional y selección de clones superiores de Guadua angustifolia Kunth en la eco-región cafetera de Colombia. Tesis doctoral. Universidad Nacional de Colombia sede Palmira. 151 p.
Nei, M. 1973. Analysis of genetic diversity in subdivided populations. Proc Natl. Acad. Sci. 70(12):3321-3323.
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics. 89:583-590.
Ochoa, E.I; Villegas, V.E y Beebe, S.E. 1997. Identificación de marcadores moleculares RAPD asociados con la resistencia al Complejo Pudrición de Cogollo en palma de aceite (Elaeis guineensis Jacq.). Revista Palmas, Vol 8, No. 2, págs: 33-38.
Ortega, D. S. 2013. Diversidad Genética y Selección en Palma de Aceite (Elaeis oleífera e Elaeis guineensis) en el Ecuador. Tesis Doctoral. Universidad Federal de Vicosa. Brasil. 51 p.
Ott, J. 1992. Strategies for chacaracterizing highly polymorphic markers in human gene mapping. Am. J. Hum. Genet. 51:283-290.
Palacio-Mejía, J. D. 2004. Estudio comparativo de la diversidad genética y divergencia evolutiva entre la especie vulnerable Colombobalanus excelsa (Lozano et al.) Nixon & Crepet y el roble común Quercus humboldtii Bondpl: implicaciones para la biología de la conservación. Tesis de maestría. Universidad Nacional de Colombia Sede Palmira. 108 p.
Peakall, R. y Smouse, P. 2012. GenAlex 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. In: Bioinformatics 28:2537-2539.
Piñero, D.; Barahona, A.; Eguiarte, L.; Rocha-Olivares, A. y Salas-Linaza, R. 2008. La variabilidad genética de las especies: aspectos conceptuales y sus aplicaciones y perspectivas en México. Capital natural de México, I: Conocimiento actual de la biodiversidad (CONABIO) ISBN: 978-607-7607-03-8. 415-435 p.
Posso, A. M. 2011. Diversidad genética y estructura poblacional de guadua angustifolia Kunth en el eje cafetero Colombiano. Tesis de maestrías. Universidad Nacional de Colombia, Sede Palmira. Palmira, Valle, Colombia. 98 p.
Purba, A. R.; Noyer, J. L.; Baudouin, L.; Perrier, X.; Hamon, S. Lagoda, P. J. L. 2000. A new aspect of genetic diversity od Indonesian oil palm (Elaeis guineensis Jacq.) revealed by isoenzyme and AFLP markers and its consequences for breeding. Theoretical Applied Genetics (Alemania). 101:956-961.
Rocha, P.J. 2004. Conceptos básicos en biotecnología de la palma de aceite. Palmas (Colombia) 25(especial):11-17.
Rocha, P.; Meléndez, E. R. y Rey, L. 2007. Ampliación del análisis de diversidad genética de palma de aceite proveniente de Angola. Respuestas. 12(1):20-28.
Shah, F. H.; Rashid, O.; Simons, A. J.; Dundson, A. 1994. The utility of RAPD markers for determination of genetic variation in oil palm (Elaeis guineensis). Theoretical Applied Genetics (Alemania) 89:713–718.
Singh, R.; Noorhariza, M. Z.; Ting N. C.; Rozana, R.; Tan, S. G. y Low E. T. L. 2008. Exploiting an oil palm EST database for the development of gene-derived SSR markers and their exploitation for assessment of genetic diversity. Biologia 63(2):227-235.
Weir, B. 1996. Genetic Data Analysis II: methods for discrete population genetic data. Sunderland: Sinauer Associates.
Weising, K. 1995. DNA fingerprinting in plants DNA fungi. CRC Press. 342 p.
Wright, S. 1931. Evolution in Mendelian population. Genetics, 16:97-159.
Wright, S. 1951. The genetical structure of populations. Annals of Eugenics. 15:323-354.
Wright, S. 1978. Evolution and the Genetics of Populations, Variability Within and Among Natural Populations. The University of Chicago Press, Chicago.
Zaki, N. M.; Ismail, I.; Rosli, R.; Chin, T. N. & Singh, R. 2010. Development and characterization of Elaeis oleifera Microsatellite Markers. Sains Malaysian. 39(6).
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
CrossRef Cited-by
1. Alina Camacho-Villalobos, Fernando Serna, Jhofre Flores, Hector Flores, Paulo Manrique, Jorge Bendezu, Tzen-Yuh Chiang. (2021). Morphological and molecular characterization of an Elaeis oleifera (H.B.K) Cortes germplasm collection located in Ucayali, Peru. PLOS ONE, 16(5), p.e0250445. https://doi.org/10.1371/journal.pone.0250445.
2. Christian Camilo Castañeda Cardona, Yacenia Morillo Coronado, Ana Cruz Morillo Conronado, Iván Ochoa. (2018). Genetic diversity in oil palm (Elaeis guineensis Jacq) using RAM (Random Amplified Microsatellites). Bragantia, 77(4), p.546. https://doi.org/10.1590/1678-4499.2017385.
Dimensions
PlumX
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2016 Acta Agronómica

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-SinDerivadas 4.0.
Política sobre Derechos de autor:Los autores que publican en la revista se acogen al código de licencia creative commons 4.0 de atribución, no comercial, sin derivados.
Es decir, que aún siendo la Revista Acta Agronómica de acceso libre, los usuarios pueden descargar la información contenida en ella, pero deben darle atribución o reconocimiento de propiedad intelectual, deben usarlo tal como está, sin derivación alguna y no debe ser usado con fines comerciales.